无缝克隆技术是时下最流行的质粒构建技术,几乎每个分子生物学实验室都在使用。以下将介绍Gibson Assembly的实验设计和操作流程,以及另外一种无缝克隆方法——Golden Gate Assembly,可以对比两种方法的优缺点和区别,希望能对大家的质粒构建实验有所帮助。
Gibson Assembly
过去的十年间,科学家开发了许多不同的克隆方法来代替限制酶克隆,这些新的方法越来越常用,相较于传统的限制克隆有许多优势。
01简介
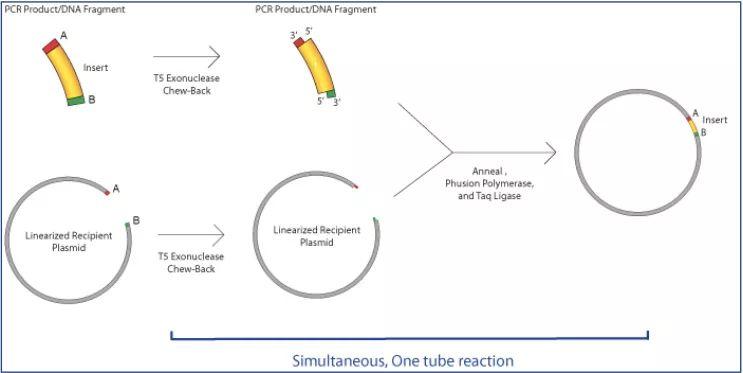
Gibson组装技术在2009年首次被Dr.Daniel和其同事提出。Gibson连接因能轻易地组装多个线性DNA片段而著名,也可以用于将一个目的片段插入选择的载体中完成基本克隆。
简单地说,进行Gibson连接时首先需要获得末端含有同源区域的DNA片段,一般是通过PCR获得,然后这些片段和酶混合物(an enzyme master mix)共孵育完成连接,该酶混合物含有三个不同的酶:
T5 核酸外切酶:用于消化片段的5’末端,产生长突出末端,允许同源单链区域退火。
Phusion聚合酶:用于填充缺口。
Taq连接酶:用于连接切口。
该酶混合物的一个优点是它们可以在同一温度下工作,所以整个反应在50℃孵育1h或更少的时间即可完成。1h以后,连接产物立即被转化入感受态细胞中。酶混合物可在生物科技公司如NEB或SGI-DNA购买,也可以自己混合。
02 Gibson连接过程
Gibson连接步骤:
- 设计引物:相邻片段的同源区域是通过使用含合适同源序列的引物进行PCR扩增添加上去的。NEB推荐使用15-40bp的重叠区,引物融解温度要高于48℃。Snapgene和NEB都有引物设计工具,可帮助设计含同源区域的引物。
- 扩增片段:可以通过PCR扩增片段和载体,鉴定片段大小并纯化DNA片段;
- 连接反应:只需要混合DNA片段、线性质粒和Gibson assembly master mix,50℃孵育1h后,将连接产物转化到感受态细胞中。
连接反应过程:
- T5核酸外切酶以5’到3’的方向消化DNA片段,每条DNA链形成一个单链DNA突出末端。
- 两相邻片段的突出末端含同源区域,DNA片段可退火,互补序列配对。
- 配对后仍存在缺口,利用DNA聚合酶补全缺口。
- 最后DNA连接酶在切口处形成磷酸二酯键,完成连接。
反应示意图如下:
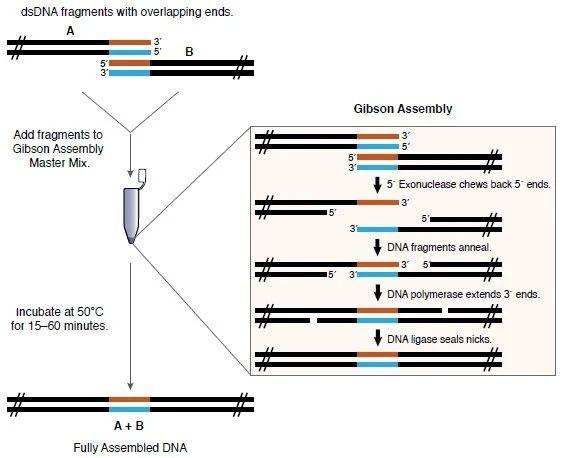
该反应的成功率非常高,一般不需要筛选大量的单克隆。除去获取引物的时间,可以在5天内完成克隆。
实验安排:
第一天:摇菌
第二天:收菌,提基因组,扩片段,胶回收;提质粒,酶切或PCR扩增获取线性质粒,胶回收
第三天:连接,转化
第四天:挑取单克隆进行PCR鉴定,取鉴定结果为成功的单克隆摇菌
第五天:收菌,提质粒,PCR、酶切鉴定,鉴定结果为成功后送测序并保菌
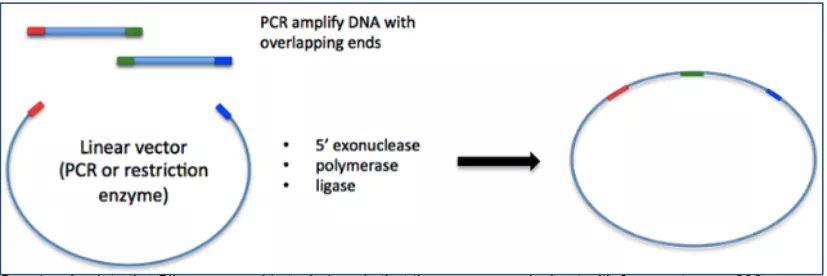
03优缺点
优点:
可一步完成多达6个片段的连接
无痕连接
相比其它克隆方法利用片段间较短的重复区域,Gibson连接利用的片段间的长重复区域更特异性地确保了连接顺序。
缺点:
连接的片段大小最好大于200bp。这可能是因为在退火和聚合步骤之前,核酸外切酶能完全消化小于200bp的片段。
如果片段的末端含有稳定的单链DNA二级结构如发夹结构或茎环结构(可能发生在终止子序列上),因为抑制了单链退火,阻止了相邻片段的连接,会导致连接效果不好。
每个片段的长引物必须专门设计,对于每个片段及其相邻片段都是特异性的,所以只能在某一特定的实验中使用。
04 结合CRISPR技术使用
Gibson连接能用于更复杂的克隆计划,比如载体片段很大,GC含量高,含有许多重复序列的克隆,以上任何一点都会使PCR步骤变得困难;又比如载体上无限制性位点帮助质粒线性化的克隆。这些情况下可以结合CRISPR技术实现实验目的。
最新研究案例显示,结合Gibson Assembly和CRISPR技术在基因组编辑方面取得了重要进展。研究表明,CRISPR技术的高效性和简便性使其成为主流,但也有科学家在探索其他基因组编辑技术。例如,南京大学团队开发了结构引导的内切酶FEN1基因组编辑系统,展示了新型编辑技术的潜力1。此外,CRISPR系统的Cas9蛋白与特定的DNA结合并切割DNA,已广泛应用于基因研究、疾病模型建立和治疗研究2。另一项研究指出,CRISPR技术在微生物合成生物学中得到广泛应用,为微生物底盘细胞构建和微生物天然产物生物合成提供了支持3。综合来看,结合Gibson Assembly和CRISPR技术在基因组编辑和微生物合成领域展现出巨大潜力,并为未来研究提供了新的可能性。
结合Gibson Assembly和CRISPR技术的研究案例包括:
- 南京大学团队开发了结构引导的内切酶FEN1基因组编辑系统,展示了新型编辑技术的潜力2
- 韩春雨团队发现来源于格氏嗜盐碱杆菌的Argonaute蛋白可以利用磷酸化的ssDNA引导切割特定的双链DNA,可在哺乳动物细胞中进行基因组编辑操作2
- CRISPR技术在微生物合成生物学中得到广泛应用,为微生物底盘细胞构建和微生物天然产物生物合成提供支持5
结合Gibson Assembly和CRISPR技术的组合可用于研究疾病,特别是基因组编辑方面。通过这种组合,可以实现精准的基因编辑和调控,有助于研究疾病相关基因的功能和影响。其中,CRISPR文库是一个基于CRISPR/Cas9技术的高通量基因筛选方案,可用于功能性筛选、富集以及深度测序分析,从而挖掘与特定表型相关的基因或筛选药物新靶点2。此外,结合Gibson Assembly和CRISPR技术可以实现高效的基因组编辑操作,例如利用内切酶FEN1基因组编辑系统进行精准编辑[1](https://www.agilent.com/cs/library/brochures/CRISPR Brochure 5991-9158ZHCN 6.0.pdf)。这种组合技术在疾病研究中具有巨大潜力,可以帮助科学家更好地理解疾病发生机制、寻找治疗方法以及开发新的治疗策略。
05 其它基于同源的克隆技术
之前我们已经介绍过不依赖于序列和连接的克隆(SLIC)。虽然SLIC相比Gbison连接更便宜,但Gibson连接也改善了SLIC方法以下的两个方面:
一:Gibson连接使用了一种专用的5’核酸外切酶而不是T4DNA聚合酶的核酸外切酶活性,不需要受dNTPs存在情况的调控。
二:Gibson连接使用的连接酶可体外修复切口,而SLIC是利用大肠杆菌的同源重组系统在体内修复载体,效率较低。
除了SLIC和Gibson,还有许多其它的基于同源重组的连接方法,如CPEC(circular polymerase extension cloning)和SLiCE(Seamless Ligation Cloning Extract)等。同样地,也有许多基于长重叠区域的克隆试剂盒,不需要限制酶,且为无痕连接。
06区分几种克隆方法
有两种常用的克隆方法经常和Gibson连接混淆,但其实工作机制完全不同:
Golden gate克隆为无痕连接,但它是利用了Type IIS 限制性内切酶切割识别位点外的DNA序列的特点,需要确保载体和DNA片段上的识别位点在正确的位置且不存在于插入片段内部。
Gateway克隆利用了 整合酶催化att位点重组,这个方法需要特异性载体,其上含有整合位点并且会在片段之间留下痕迹,但是该方法可以轻易地将DNA片段从一个载体上移到另一个载体上。
现在有许多不同的克隆方法。Gibson和其它的基于长同源序列的克隆方法是替代限制克隆、Gateway或者Golden gate克隆的一种好方法。不管是要常规克隆、多片段克隆还是用于合成生物学,都应该尝试着使用一下该方法。
Golden gate
Golden gate 的方法跟传统的分子克隆的方法的优势相比在于可以实现片段的边切变边连,节省时间,同时可以连个多个片段的连接。
Golden Gate Assembly是一种新型的酶切连接方法 [1, 2],与传统的酶切连接方法不同。传统的酶切连接方法采用标准的II型限制性内切酶 (限制酶) (例如EcoRI) 切割DNA,这些限制酶通常识别4~8 bp的回文序列,并在识别序列内部切割产生粘性末端或平末端 (图1A)。而Golden Gate Assembly采用IIS型限制酶 (例如*Bsa*I) 切割DNA,这些限制酶识别非回文序列,并在识别序列外部切割产生粘性末端 (图1B)。
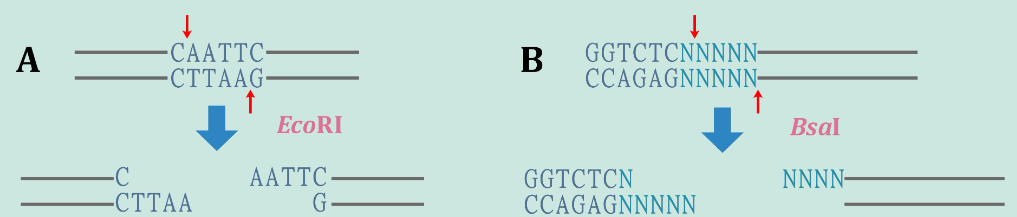
IIS型限制酶切割DNA产生的粘性末端通常为2个碱基或4个碱基。在连接反应中,粘性末端的碱基数越多,连接产物的保真度越高,因此Golden Gate Assembly通常采用能够产生4碱基粘性末端的IIS限制酶,**常用的有*Bsa*I、*Bsm*BI和*Bbs*I (图2)。NEB公司对这三种野生型的限制酶进行了分子改造,得到三种识别序列不变但特异性更强的限制酶,分别是*Bsa*I-HFv2、*Bsm*BI-v2和*Bbs*I-HF (图2)**。这些限制酶的识别序列长度都是6 bp,因此在DNA序列中出现的概率都是1/4096。
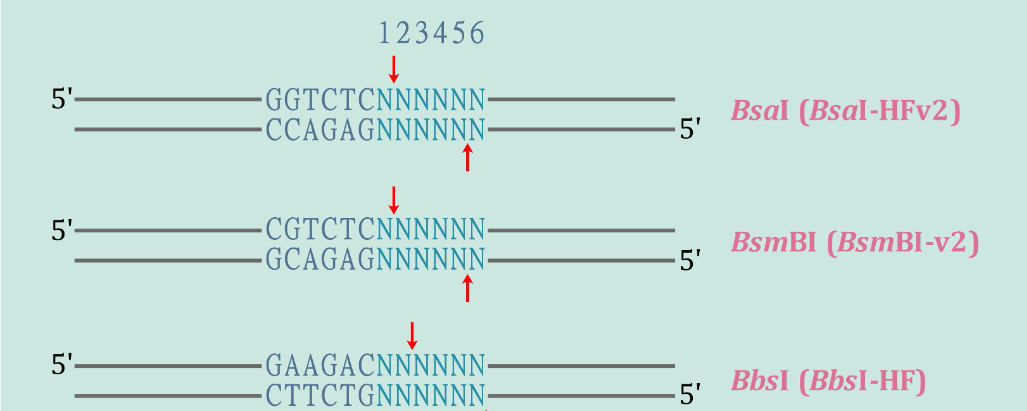
如图2所示,*三种IIS型限制酶的特异性可分别记为:*Bsa*I (GGTCTC 1/5),*Bsm*BI (CGTCTC 1/5),*Bbs*I (GAAGAC 2/6)*。除此之外,能够产生3碱基粘性末端的IIS型限制酶Sap*I (GCTCTTC 1/4) 也常被使用 [3],这个限制酶虽然产生的粘性末端更短,导致连接产物的保真度会略有降低,但是识别序列长度更长 (为7 bp),因此在DNA序列中出现的概率是1/16384。根据切割位点序列的不同,BsaI、BsmBI和BbsI可以产生的粘性末端有256种,Sap*I可以产生的粘性末端有64种。
市面上有许多基于Golden Gate Assembly的克隆试剂盒,不过价格都很贵,我一向建议大家自己买酶和试剂配制反应体系,只要弄懂Golden Gate Assembly的工作原理和实验设计方法就没有问题。下面我以BsaI-HFv2为例,讲讲如何使用Golden Gate Assembly构建质粒。
- 设计引物
通过PCR引入BsaI的识别序列,识别序列加在引物的5’端,为了确保限制酶能稳定结合到DNA双链上并发挥切割作用,需在识别序列末端加上保护碱基。保护碱基的数量和种类不是固定的,为了简化引物设计,大家可以用TTT作为保护碱基(3 bp的保护碱基对于绝大多数限制酶来说是足够的,若是为了保险起见,也可将保护碱基增加至6 bp。由于切割位点在识别序列的下游且可以是任意序列,因此有3种不同的引物设计方法,如图3所示。
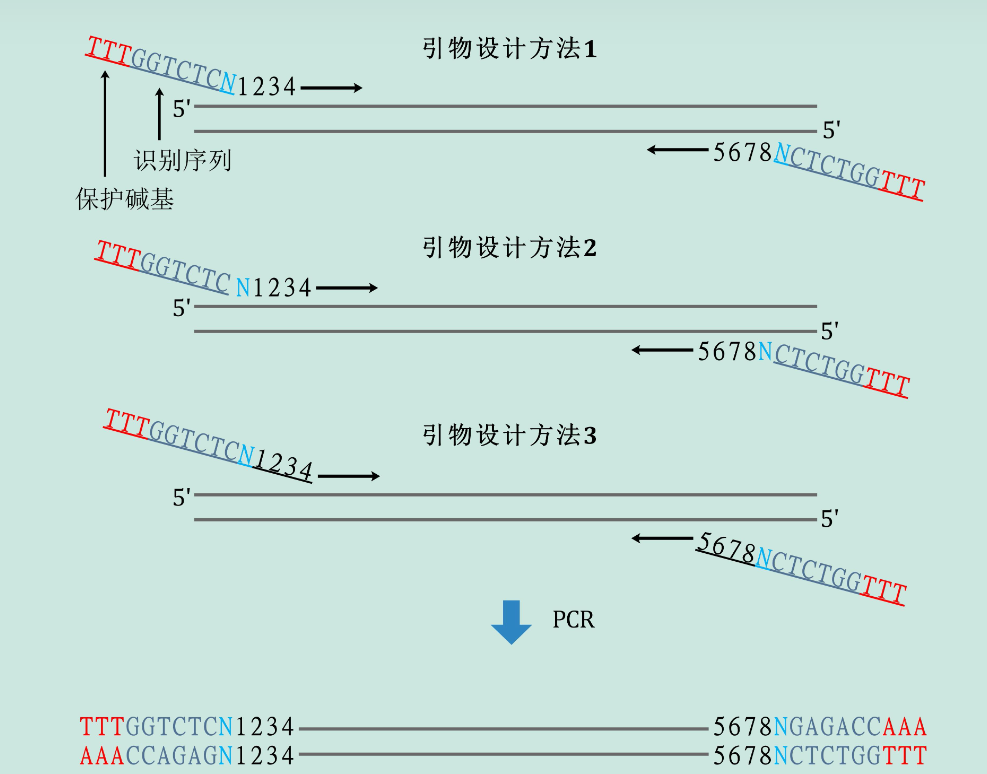
[^图3]: 设计引物的三种策略:N代表任意碱基, 1234和5678代表两个不同的切割位点, N和切割位点既可以利用PCR模板现有的序列,也可以通过引物引入
- 实验原理
(1) 2个DNA片段的连接
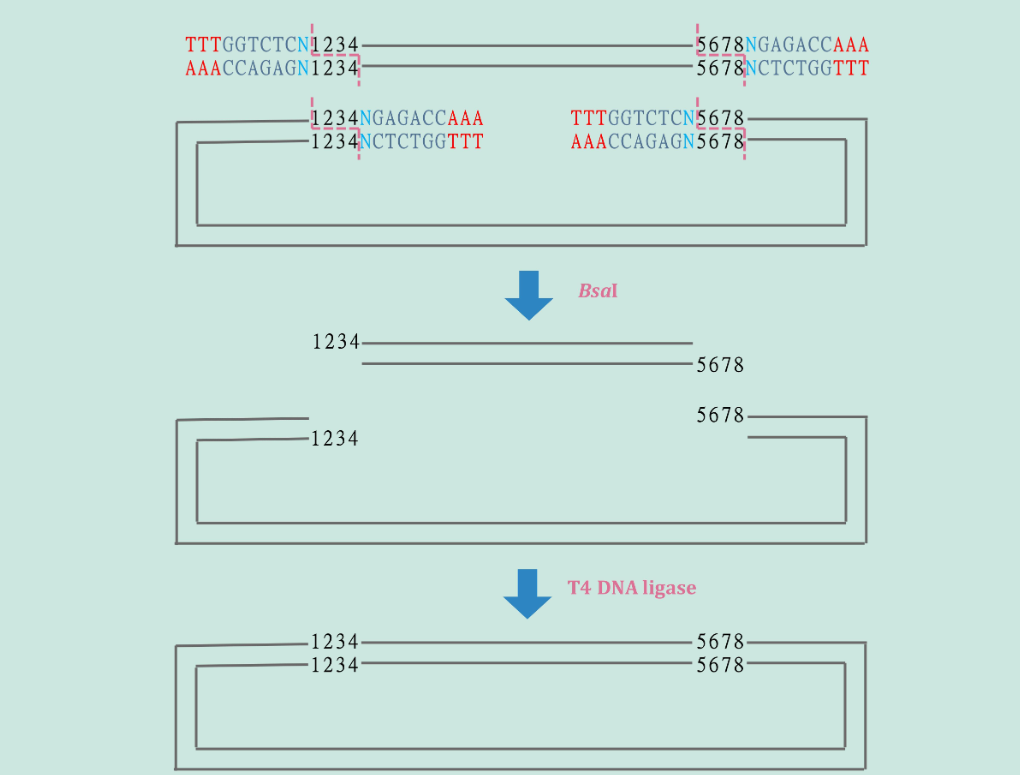
[^图4]: Golden Gate Assembly连接两个片段
(2) 4个DNA片段的连接
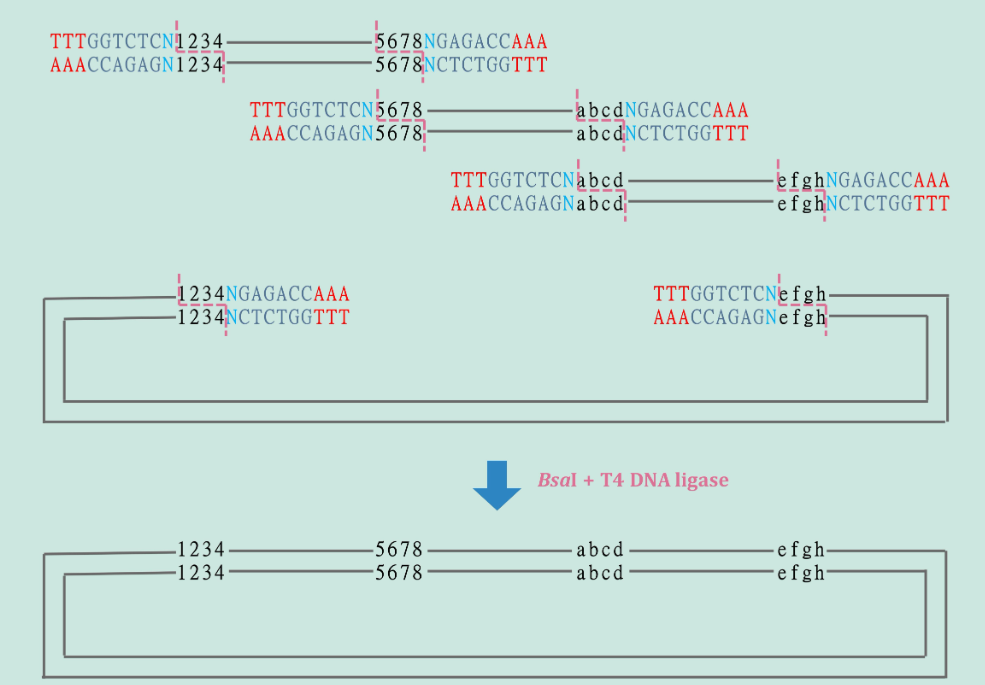
[^图5]: Golden Gate Assembly连接四个片段:1234, 5678, abcd和efgh代表四个不同的切割位点
上面两个图分别展示了2个片段连接和4个片段连接的工作原理,其他数量片段的连接类似。酶切和连接是在同一个反应体系中进行的,可以用PCR产物作为底物 (如图4和图5),也可以先将PCR产物分别克隆到质粒载体上,再用质粒作为底物。在合适的实验条件和实验设计下,利用Golden Gate Assembly可以组装超过20个DNA片段。下面这个小视频可以帮助大家进一步理解Golden Gate Assembly的工作原理。
- 操作流程
(1) 配制反应体系
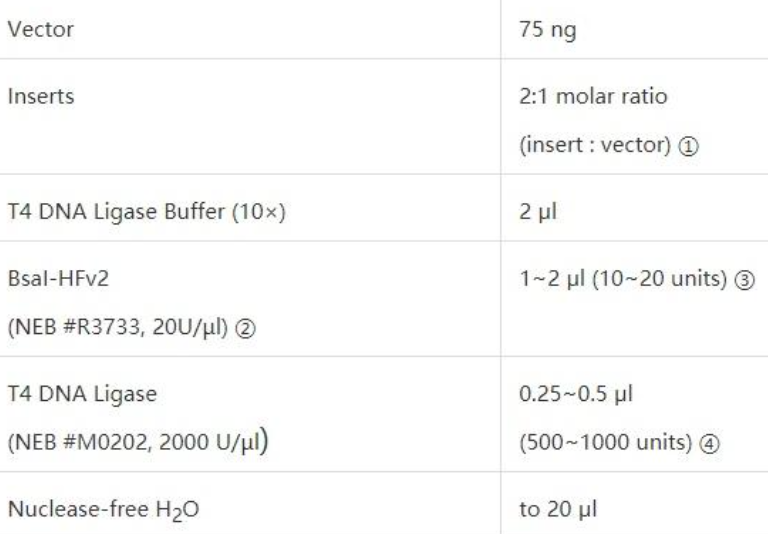
① 每个插入片段的加入量可根据载体的加入量与载体和插入片段的长度进行换算,这里提供一个换算工具http://nebiocalculator.neb.com/#!/ligation; ②其他可用的内切酶:BsmBI-v2(NEB #R0739, 10U/µl), BbsI-HF (NEB #3539, 10U/µl), SapI (NEB #R0569,10U/µl); ③若插入片段数量为110,则用10 units;若插入片段数量大于10,则用20 units; ④ 若插入片段数量为110,则用500 units;若插入片段数量大于10,则用1000units。
(2) 反应程序
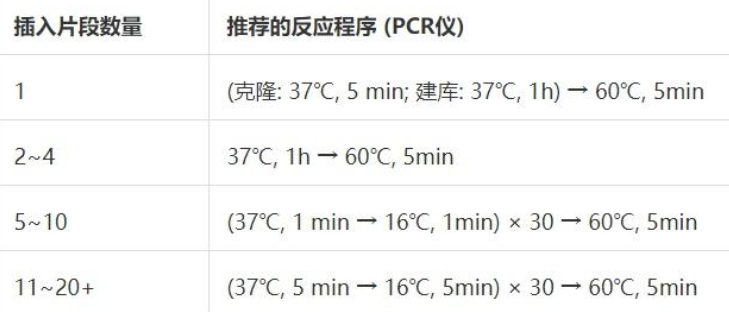
注: 37℃是BsaI-HFv2的最适反应温度,若使用其他限制酶,该温度可能需要调整,比如BsmBI-v2的最适温度为42℃。
- 优点
与传统的酶切连接比较
(1) IIS型限制酶的切割位点在识别序列外部,因此识别序列在酶切过程中被除去,不会在连接产物中引入不必要的序列。
(2) IIS型限制酶对切割位点的序列没有要求,因此可以通过设计切割位点的序列产生不同的粘性末端,使得多片段定向组装成为可能。
(3) 由于识别序列不会残留在连接产物中,因此酶切反应和连接反应可以在同一个反应体系中进行,简化了实验流程。
与Gibson Assembly比较
(1) 片段连接过程中不涉及序列的降解和合成,因此连接产物的保真度更高。
(2) 可以组装的DNA片段数量更多,适用于质粒建库。
(3) 可以组装带有同源序列或重复序列的DNA片段,比如可用于构建表达多个sgRNA的质粒 [4]。
(4) 可以构建标准化的合成生物学元件,这一点类似于和BioBricks。
- 注意事项
(1) 需选用合适的限制酶,该限制酶的识别序列在最终的连接产物中不能出现。
(2) 设计引物时,识别序列和切割位点的取向要正确。
(3) 当插入片段的数量较多时,为了确保连接的特异性,需要合理设计切割位点的4 bp序列。
[1] Engler, C., et al. (2008) PLoS One 3, e3647.
[2] Engler, C.,et al.(2009) PLoS One 4, e5553.
[3] Whitman, L., et al. (2013) Genet. Eng. Biotechnol. 33, 42.
[4] Vad-Nielsen, J., et al. (2016) Cell Mol. Life Sci. 73, 4315–4325.
https://doi.org/10.1007/s00018-016-2271-5doi.org/10.1007/s00018-016-2271-5
[5] HamediRad, M., et al. (2019) ACS Synth. Biol. 8, 1047–1054.
Highly Efficient Single-Pot Scarless Golden Gate Assemblydoi.org/10.1021/acssynbio.8b00480
- 本文作者: Anderson
- 本文链接: http://nikolahuang.github.io/2024/03/12/Golden gate和Gibson Assembly技术有什么区别?/
- 版权声明: 转载请注明出处,谢谢。

