G Camille M. Le Gall *,FF Felix L. Fennemann *,JS Johan M.S. van der Schoot *,FS Ferenc A. Scheeren ,MV Martijn Verdoes
,
第一次发布: 2023年02月20日第13卷第4期 DOI: 10.21769/BioProtoc.4613
评审: Luis Alberto Sánchez VargasDay-Yu Chao 匿名评审
Abstract
Köhler和Milstein在1975年开发的杂交瘤技术彻底改变了免疫学领域,使得单克隆抗体(mAbs)在研究和开发中得以常规使用,并最终在临床上成功应用。尽管重组生产需要符合良好生产规范的技术来生产临床级别的mAbs,但学术实验室和生物技术公司仍然依赖原始的杂交瘤细胞系,以稳定、轻松地以适中价格生产高产量抗体。在我们的工作中,使用杂交瘤来源的mAbs时遇到了一个主要问题:无法控制生产的抗体格式,而重组生产则允许这种灵活性。为了消除这一障碍,我们通过直接在杂交瘤细胞的免疫球蛋白(Ig)位点进行基因工程改造抗体。我们使用成簇规律间隔短回文重复(CRISPR)/CRISPR相关蛋白9(Cas9)和同源指导修复(HDR)来修改抗体的格式[mAb或抗原结合片段(Fab’)]和抗体同种型。本方案描述了一个简单的方法,操作时间少,能够产生稳定分泌高水平工程抗体的细胞系。保持亲本杂交瘤细胞在培养中,转染针对Ig位点中兴趣位点的导向RNA(gRNA)和HDR模板以敲入所需插入物和抗生素抗性基因。通过施加抗生素压力,扩大抗性克隆,并在基因和蛋白质水平上对其是否能产生修改后的mAbs而非亲本蛋白进行表征。最后,在功能分析中表征修改后的抗体。为了展示我们策略的多样性,我们用以下例子来说明本方案:一是更换抗体的恒定重区,创建新型同种型的嵌合mAb;二是截断抗体以创建抗原肽融合的Fab’片段,以产生针对树突细胞的疫苗;三是修改重链(HC)的恒定重(CH)1域和轻链(LC)的恒定kappa(Cκ)以引入用于进一步衍生纯化蛋白的位点选择性修饰标签。只需使用标准的实验室设备,这有助于其在各种实验室的应用。我们希望这个方案能够进一步传播我们的技术,帮助其他研究人员。
背景介绍:
使用瞬时重组系统生产抗体是劳动密集型的,并且由于可变的转染效率和需要抗体可变区序列的知识,面临着可重复性问题。这对于拥有专用生物工厂的专业公司和实验室来说是常规操作,但典型的学术实验室通常不具备这些基础设施。对于大多数实验室来说,杂交瘤是生产单克隆抗体(mAbs)最便宜且劳动强度最低的方法。
由于最初细胞系产生的免疫动物经历了多轮免疫和类别转换重组,同种型之间存在着微小的多样性,几乎全部是免疫球蛋白(Ig)G亚型。尽管同种型并不总是相关的,但它对于治疗的有效性越来越明显(Waldor等人,1987年;Beers等人,2016年;Evers等人,2021年);例如,在癌症治疗中使用的mAbs的同种型工程可以显著提高其疗效(Sharma等人,2019年)。抗原结合片段(Fab’)由于其小尺寸,在研究中是宝贵的工具,但需要使用木瓜蛋白酶对抗体进行后隔离修改(Zhao等人,2009年)。最后,抗体的化学修饰通常是通过赖氨酸残基的随机标记来实现的(Nanda和Lorsch,2014年),这不是特定位点的,会导致不希望有的异质产品,或者通过工程化序列引入未配对的半胱氨酸残基(Adhikari等人,2020年),尽管正在开发新的方法(Yamada等人,2019年)。
为了共同解决这些问题,我们开发了一种技术,可以重复地使用成簇规律间隔短回文重复(CRISPR)/CRISPR相关蛋白9(Cas9)修改杂交瘤中的Ig恒定位点。这种现在常规的基因编辑方法能够精确地靶向Ig位点的恒定区域,无论可变区域如何,并能够插入几乎任何序列来生产具有任何切换或突变同种型、格式或标签的抗体。在这种情况下,杂交瘤中同种型的低多样性是一个优势,因为同源指导修复(HDR)模板可以轻松地在许多细胞系上重复使用。虽然我们专注于编辑恒定重链(CH)1、铰链(H)和轻链(LC)区域,但其他人已经证明了CH3靶向的可行性(Khoshnejad等人,2018年)。总的来说,这展示了策略的多样性。
我们预计这个方案可以应用于任何领域,因为mAbs是生物研究的基础,并希望看到同行成功地应用这种方法,以实现我们无法预见的用途。
实验材料与试剂
875 cm² 5层培养瓶 (康宁, Falcon®, 目录号: 353144)
75 cm²培养瓶 (Greiner Bio-one, Cellstar, 目录号: 658170)
25 cm²培养瓶 (Greiner-Bio one, Cellstar, 目录号: 690175)
10 cm培养处理的Petri盘 (Greiner Bio-one, Cellstar, 目录号: 664161)
6孔板 (康宁, Costar, 目录号: 3516)
24孔板 (康宁, Costar, 目录号: 3524)
96孔板 (康宁, Costar, 目录号: 3799)
50 mL Falcon管 (赛默飞世尔, BrandTM 114821, 目录号: 10420602)
15 mL Falcon管 (赛默飞世尔, BrandTM 114818, 目录号: 10345521)
1.5 mL管 (Eppendorf, 目录号: 0030120086)
PCR管 (Eppendorf, 目录号: 0030124820)
一次性色谱柱 (Bio-Rad, 目录号: 7321010)
pX330-U6-Chimeric_BB-CBh-hSpCas9 (Addgene, 目录号: 42230)
pSMART-HCKan, CloneSmart Blunt Cloning kit (Lucigen, 目录号: 40704-2)
pHybr_r2a>mA-srt-his (Addgene, 目录号: 124806)
pHybr_r2a>m2a(silent)-srt-his (Addgene, 目录号: 124807)
PX458-gR2A_ISO (Addgene, 目录号: 124808)
BbsI-HF® (新英格兰生物实验室, 目录号: R3539)
rSAP (新英格兰生物实验室, 目录号: M0371)
T4 DNA连接酶 (新英格兰生物实验室, 目录号: M0202)
T4 PNK (新英格兰生物实验室, 目录号: M0201)
Q5聚合酶 (新英格兰生物实验室, 目录号: M0491)
dNTP (赛默飞世尔, 目录号: R0192)
ddH2O (赛默飞世尔, 英杰生命技术, 目录号: AM9935)
琼脂糖 (Sigma-Aldrich, 目录号: A4718)
Nancy-520 (Sigma-Aldrich, 目录号: 01494)
NucleoBond Xtra Midi Plus EF kit (Macherey-Nagel, 目录号: 740422.50)
NucleoSpin Gel and PCR cleanup (Macherey-Nagel, 目录号: 740609.50)
ISOLATE II Genomic DNA isolation kit (Bioline, 目录号: BIO-52067)
杂交瘤: 大鼠IgG2a,λ抗小鼠PD-L1 [MIH5] (自备)
杂交瘤: 大鼠IgG2a,κ抗小鼠DEC-205 [NLDC-145] (ATCC, 目录号: HB-290TM)
杂交瘤: 小鼠IgG1,κ抗人CD20 [NKI.B20/1] (自备)
SF细胞系4D-NucleofectorTM X套件L (Lonza Biosciences, 目录号: V4XC-2024)
PBS (Fresnius Kabi, 目录号: M087312/01)
RPMI-1640 (赛默飞世尔, GibcoTM, 目录号: 42401018)
CD杂交瘤培养基 (赛默飞世尔, GibcoTM, 目录号: 11279023)
热灭活胎牛血清 (FBS) (HyClone, 目录号: SV35959)
Ultraglutamine-1 (Lonza Biosciences, 目录号: BE17-605E/U1)
抗生素-抗真菌剂 (赛默飞世尔, GibcoTM, 目录号: 15240062)
β-巯基乙醇 (β-ME) (Sigma-Aldrich, 目录号: 444203)
台盼蓝 (赛默飞世尔, GibcoTM, 目录号: 15250061)
CELLine CL 1000生物反应器 (INTEGRA Biosciences AG, 目录号: 90005)
博来霉素 (Invivogen, 目录号: ant-bl-05)
嘌呤霉素 (Invivogen, 目录号: ant-pr-5)
牛血清白蛋白 (BSA) (Sigma-Aldrich, 目录号: A9418)
NaN3 (Sigma-Aldrich, 目录号: 26628-22-8)
重组蛋白G (赛默飞世尔, PierceTM, 目录号: 21193)
Ni-NTA琼脂糖 (Qiagen, 目录号: 30230)
抗6×His标签抗体 (克隆J095G46) (PE, Biolegend, 目录号: 362603)
抗大鼠IgG2a重链 (克隆MRG2a-83) (PE, Biolegend, 目录号: 407507)
抗小鼠IgG2a (克隆m2a-15F8) (PE, eBioscience, 目录号: 12-4817-82)
兔抗6×His标签抗体 (克隆RM146, 非结合, Abcam, 目录号: AB14923)
山羊抗兔IgG (H+L) (多克隆, IRD800, LI-COR, 目录号: 926-32211)
山羊抗大鼠IgG (H+L) (多克隆, AF680, 赛默飞世尔, 目录号: A-21096)
洗涤缓冲液 (见配方)
PBA (见配方)
培养基 (见配方)
1×选择培养基 (见配方)
设备
电穿孔设备,例如 AMAXA 4D Nucleofector (Lonza BioSciences, AAF-1002X/AAF-1002B)
带5% CO2的湿化培养箱
热循环器(例如,Bio-Rad, 目录号:1861096EDU)
流式细胞仪,例如,FACSLyric (BD Biosciences, 目录号:663518)
荧光扫描仪,例如,Amersham TyphoonTM 5 (Cytiva, 目录号:29187191)
Sanger测序服务
实验步骤
A. 一般注意事项
在进行杂交瘤细胞修改之前,需要了解宿主物种和抗体同种型的信息。我们使用了剪切位点来在重链(HC)的CH1上游或下游、铰链的上游以及轻链(LC)的Cκ下游敲入插入物,但实际上几乎可以使用任何其他剪切位点来根据预期用途定制抗体。
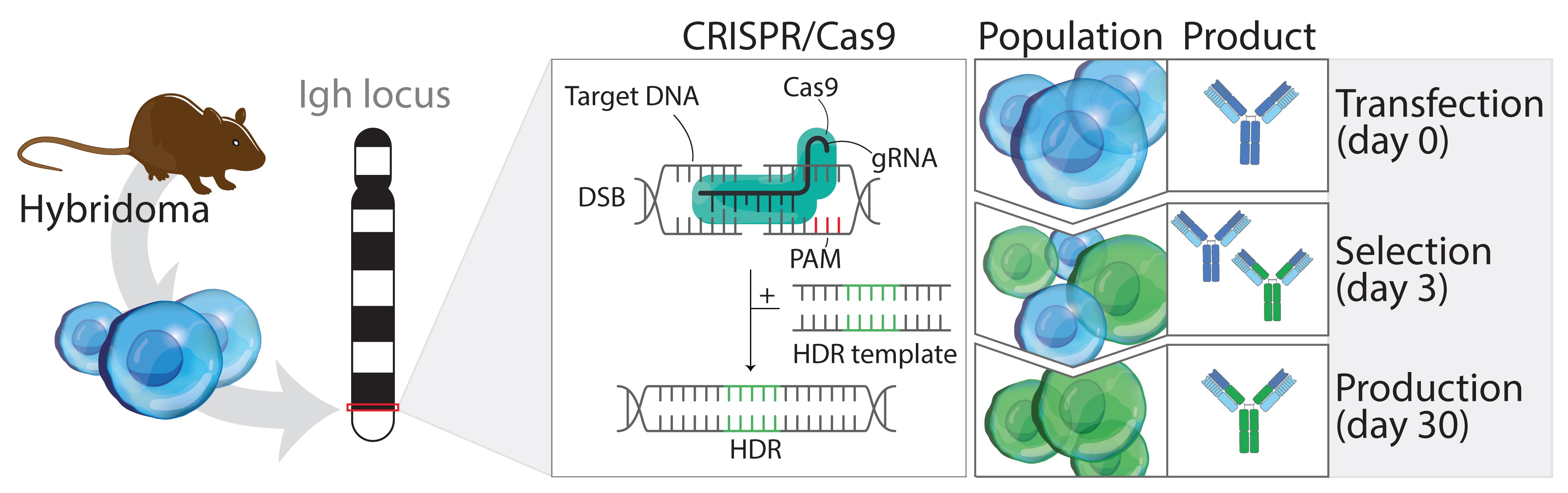
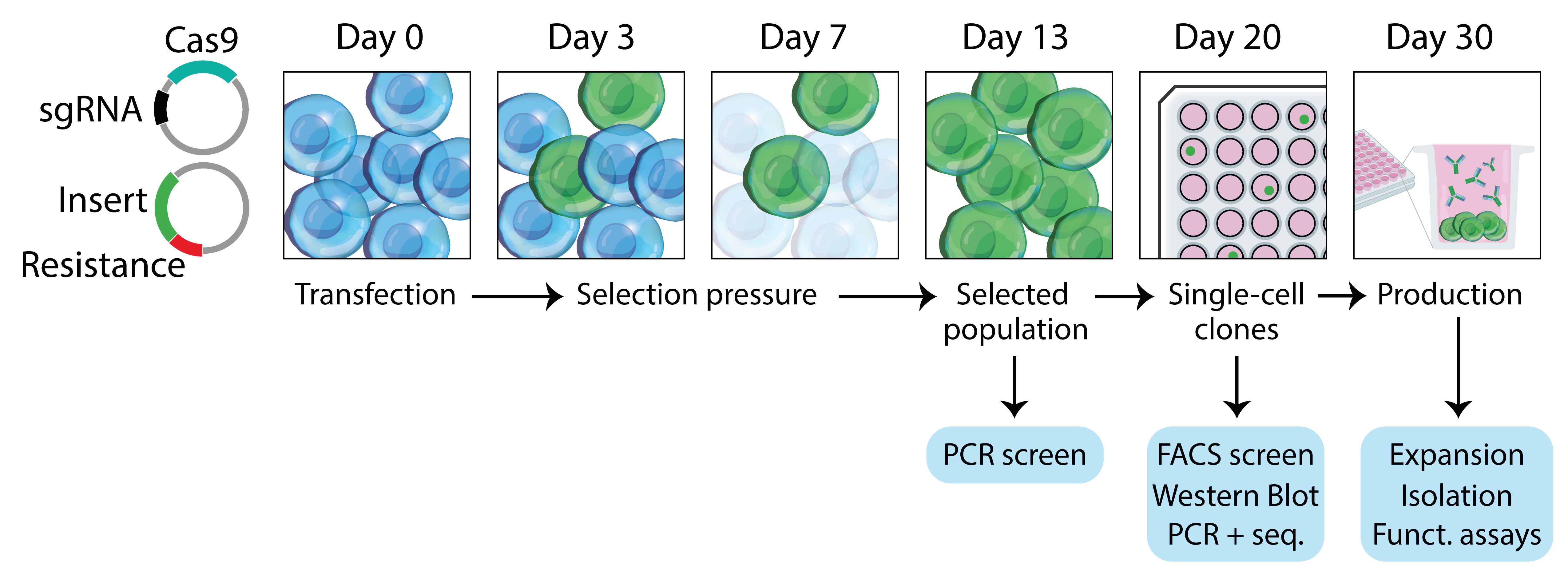
从目标细胞系的转染到高水平修改抗体(图1)的分离,需要4-6周的时间。
为了本方案的目的,并且为了展示潜在应用的多样性,我们描述了三个成功编辑的杂交瘤的全面表征:将大鼠IgG2a(r2a)抗体的恒定重链(HC)切换到小鼠IgG2a(m2a)恒定HC并产生嵌合r2a(V):m2a(C) mAb,在r2a HC的铰链区域引入抗原以产生Fab’片段-抗原融合,以及修改小鼠IgG1(m1)HC和鼠标kappa(mκ)LC以引入两个位点特异性标记标签。
B. CRISPR/HDR模板和gRNA的设计
要从头开始设计HDR模板和gRNA,Addgene CRISPR指南或CRISPR 101书籍是很好的起点。
在图2-6中,逐步展示了设计CRISPR/HDR模板和gRNAs的过程。我们以靶向m1 CH1的C端为例,工程化一个分泌mAb的细胞系,以产生在CH1 C端带有位点特异性标记位点的Fab’片段。
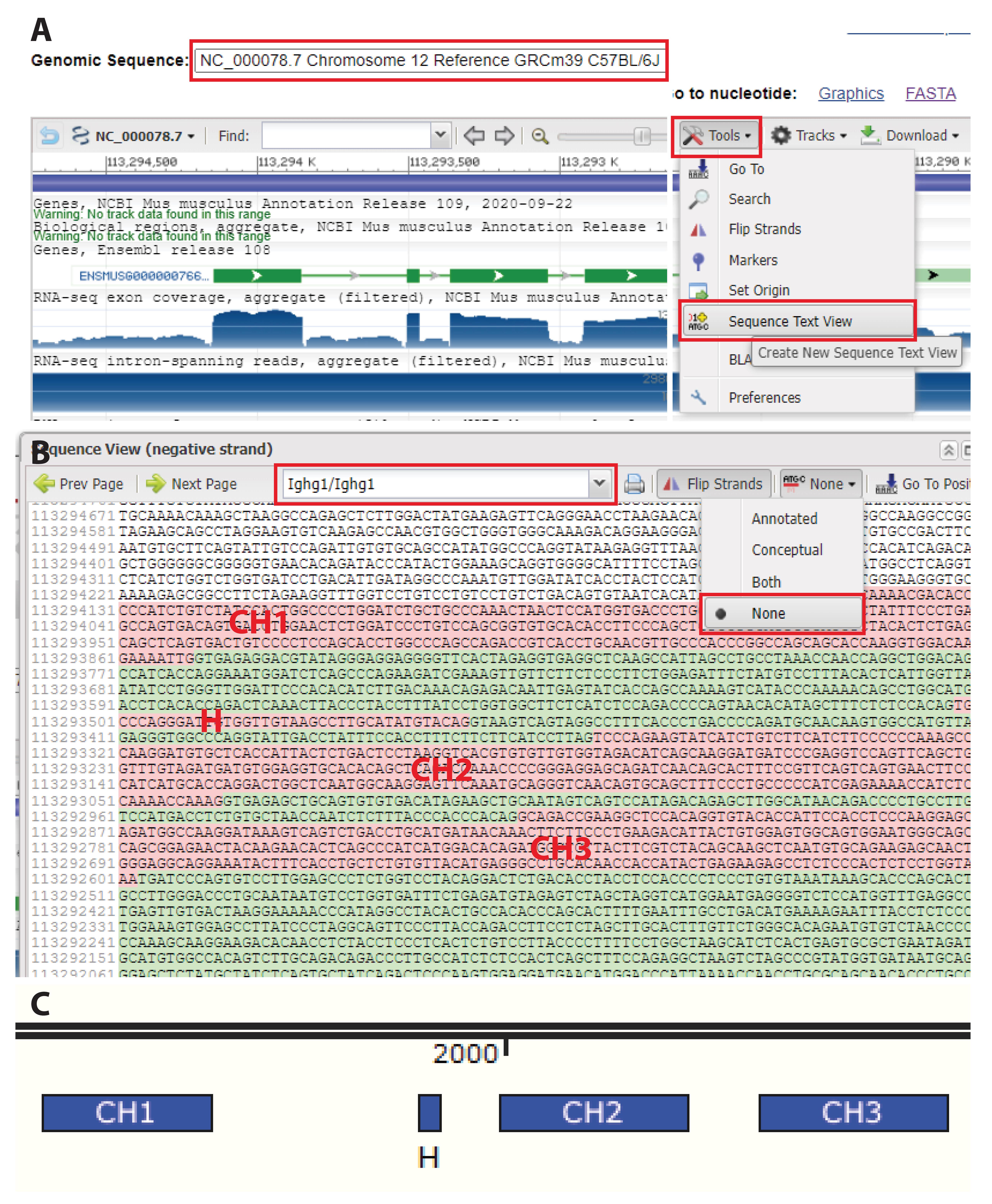
[^图2. 目标序列的注释。]: A. 访问感兴趣位点的基因组序列(例如,在NCBI基因库上)。使用“序列文本”视图,可视化目标位点的外显子和内含子。B. 移除注释,仅显示核苷酸序列。对于给定的位点,外显子以红色标记,内含子以绿色标记。在这里,高亮显示了Ighg1基因(mIgG1恒定区)中的恒定重链(CH)1、铰链、CH2和CH3。C. 在Snapgene中复制序列并注释外显子,以识别目标区域。这通常取决于预期的修改。在这里,我们目标是mIgG1 CH1的C端。
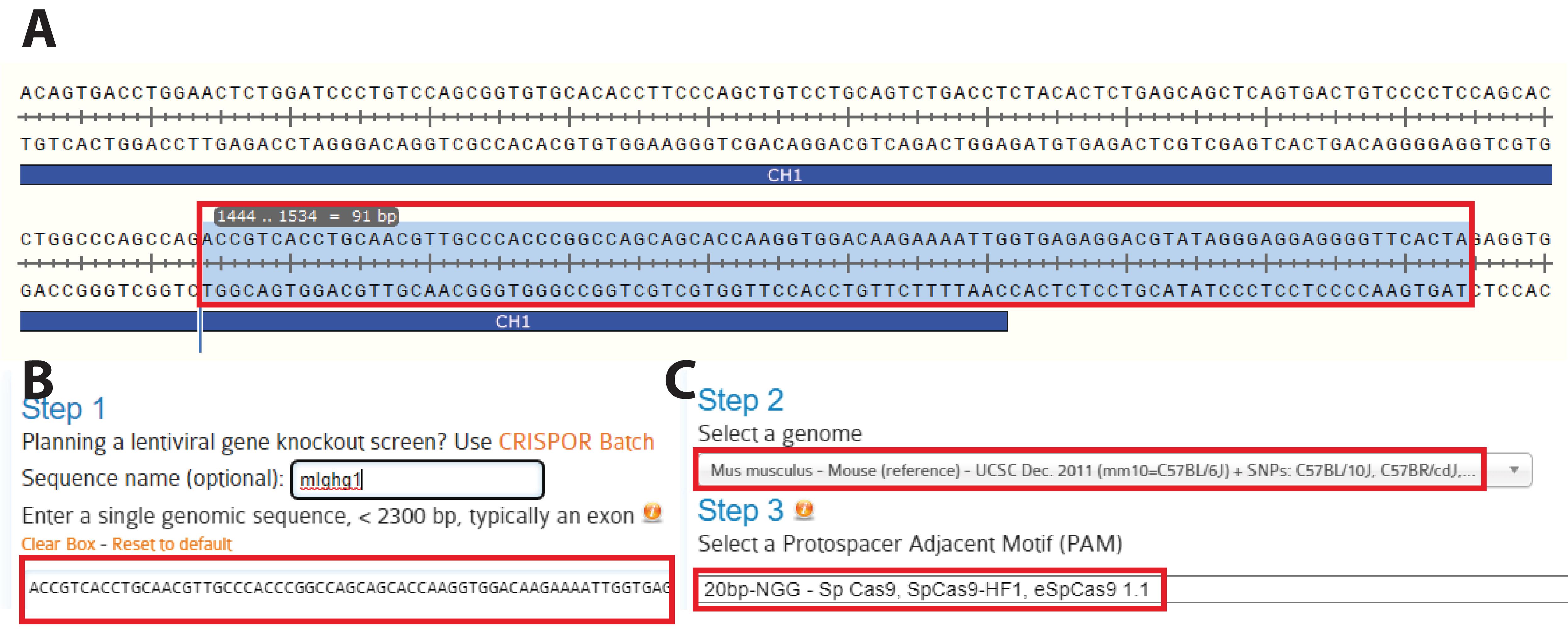
[^图3. 目标位点的识别。]: A. 确定要敲入的目标区域。在这里,我们选择CH1域的C端插入部分铰链区域、一个短连接器、一个位点特异性标记位点和一个组氨酸标签。这实际上将mAb转化为在其重链(HC)C端带有位点特异性标记位点的Fab’片段。B. 将选定的序列复制到CRISPOR。C. 选择适当的宿主(在这里,为Mus musculus)和protospacer adjacent motif(PAM)(典型为-NGG)并提交。
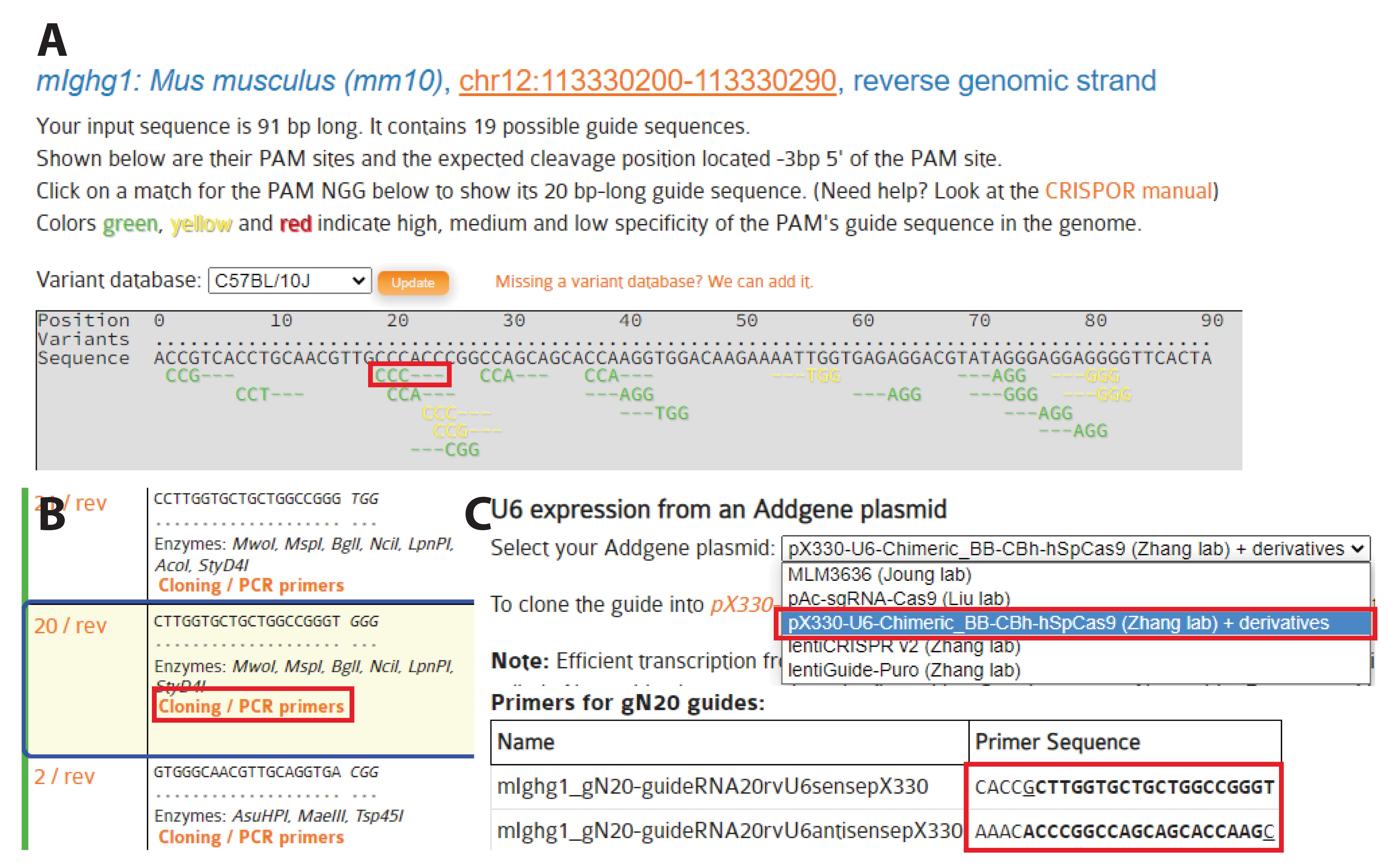
[^图4. gRNA的选择。]: A. CRISPOR识别PAM序列和相应的gRNA候选序列。所有可能的PAM序列(在这里为-NGG)都输入序列下方识别。B. 显示潜在gRNA的序列和特性,作为表格,按预测效果排序。插入位点应尽可能远离双链断裂10 bp以内。当靶向新区域时,根据其接近预期插入位点的位置和预测评分,至少选择两个或三个gRNAs。在这里,我们选择了gRNA_m1_HC(5′-CTTGGTGCTGCTGGCCGGGT-3′)。通过点击“克隆”,一个新窗口打开,显示各种Cas9载体中的克隆序列。C. 我们使用了一个U6表达系统(U6启动子,pX330载体及其衍生物)(Cong等人,2013年)。在这里,我们使用了一个20 nt的gRNA,但最近有提议使用19 nt的gRNA以提高效率(Kim等人,2020年)。选择适当的引物,并按照说明将gRNAs克隆到pX330载体中的BbsI位点。
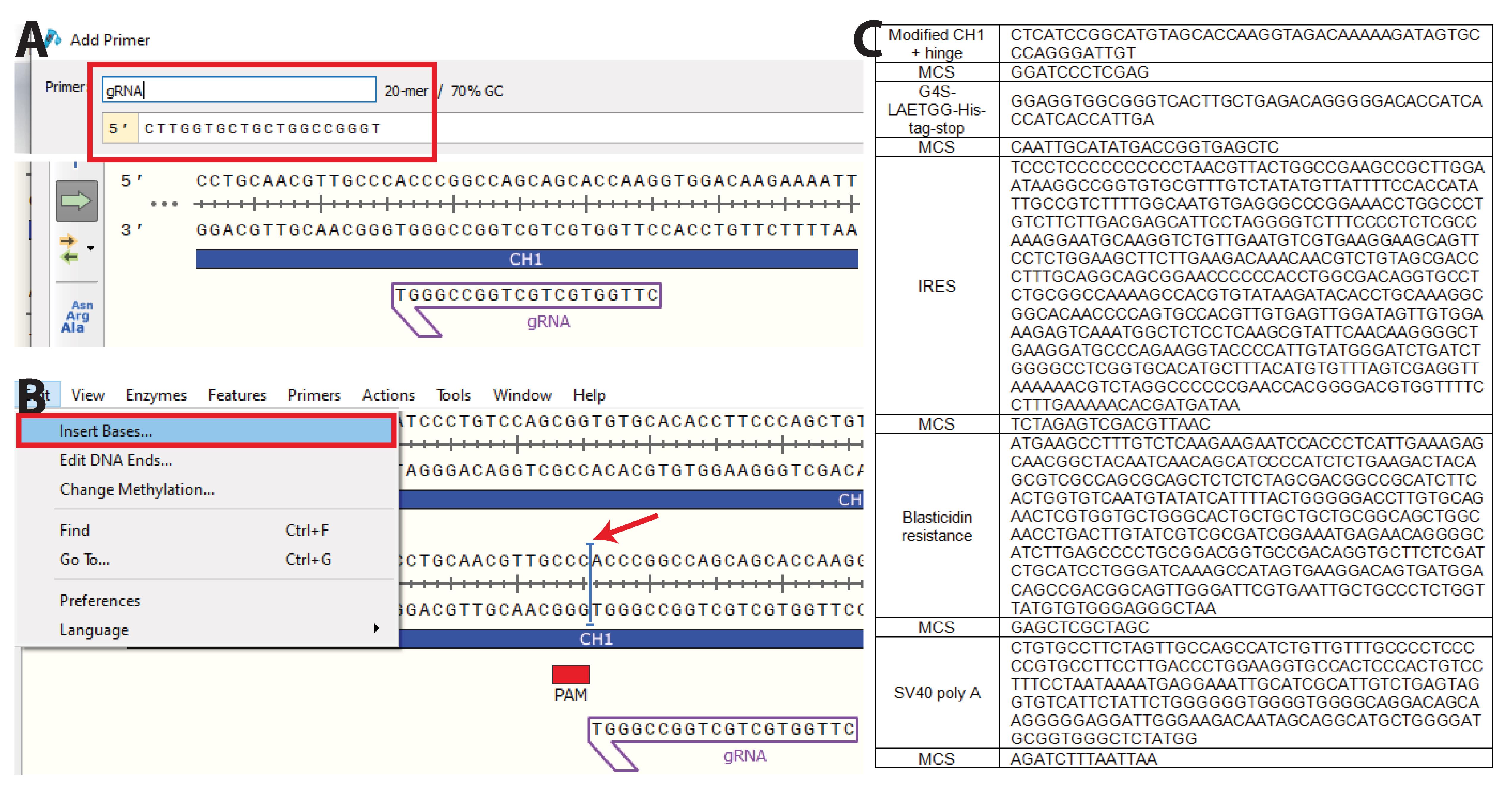
[^图5. 在敲入位点插入所需序列。]: A. 将gRNA序列(s)复制到Snapgene并B. 显示protospacer adjacent motif(PAM)序列(s)。将序列插入尽可能接近PAM序列的位置。C. 插入预定的包含所需插入物的核苷酸。在这里,我们插入部分铰链区域(VPRDC),允许与下游的轻链(LC)结合,之后是一个多克隆位点(MCS)、一个灵活的连接器(GGGGS)、一个sortag motif(LAETGG)和一个组氨酸标签(HHHHHH),之后是一个终止密码子。插入物后面是一个内部核糖体进入位点(IRES),允许转录博来霉素抗性基因(BSD),以及一个猴空泡病毒40(SV40)多聚A尾,以解离核糖体。每个域都被一个MCS包围,允许轻松交换质粒的任何部分。在这里,我们另外优化了CH1的一部分,以改善与LC的结合(PACSTKVDKKI,S>C突变)。
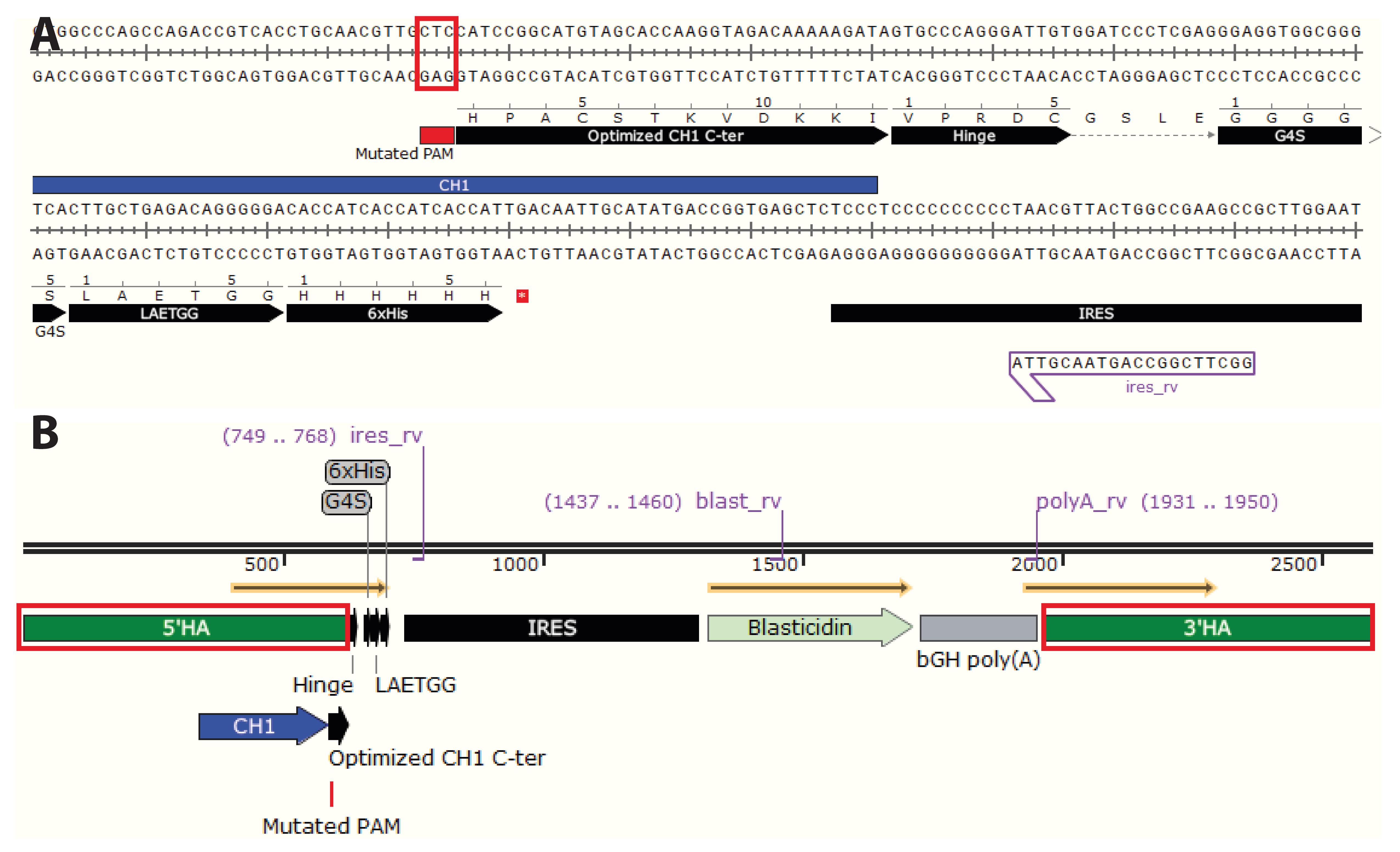
[^图6. 同源臂(HA)和最终HDR模板设计。]: 同源臂(HA)大约在插入物的每侧延伸0.6 kb。它们的大尺寸允许在同源臂和目标位点之间存在一些不匹配,而不会影响HDR过程中同源臂与基因组位点的结合。因此,与最初从基因组浏览器获得的祖源序列相比,可以在同源臂序列中进行一些小的调整,而不会影响HDR的效率。A. 需要突变“NGG” protospacer adjacent motif(PAM)序列。为了失活PAM序列,将其突变“NGG”为“NGC”、“NGA”或“NGT”。如果PAM序列位于编码区,确保它是沉默突变。或者,完全移除PAM序列。经历过成功的HDR的细胞将不包含PAM序列,Cas9将被失活。B. 为了定义5’HA和3’HA,选择插入物每侧约0.6 kb的序列。
Table 1. gRNA used or mentioned in this protocol. HA sequences for the corresponding HDR templates are reported in Table 5.
gRNA Target Sequence gRNA_iso_r2a Upstream of rat IgG2a CH1 exon TGTAGACAGCCACAGACTTG gRNA_r2a_H Upstream of rat IgG2a hinge exon GACTTACCTGTACATCCACA gRNA_m1_HC C-terminus of mouse IgG1 CTTGGTGCTGCTGGCCGGGT gRNA_mκ_LC C-terminus of mouse κ LC GGAATGAGTGTTAGAGACAA 恒定免疫球蛋白位点的序列可以在NCBI基因库中找到;PAM序列和gRNA可以使用CRISPOR或其他工具确定。
HDR模板应包含5’和3’同源臂(HAs)包围所需插入物,一个终止密码子,一个内部核糖体进入位点(IRES),一个抗生素抗性基因(博来霉素或嘌呤霉素),以及一个聚A尾(图8A-C)。插入物的长度通常为1-2 kb。虽然更长插入物是可能的,但超过2-3 kb后,HDR效率会大大降低。HAs的长度取决于插入物的长度(每1 kb插入物约0.5 kb HAs),通常在0.3 kb到1 kb之间,尽管更短的HAs也是可能的。在这里,我们使用了0.6 kb的HAs。
建议:对目标区域进行测序,以确保不存在与原始基因的偏差,并确保HDR模板中的HAs移除了PAM序列和下游外显子的剪接受体序列。表2中提供了通用测序引物,并在补充文件中提供了通用PCR协议。通过T7试验确定感兴趣区域的gRNA效率。
HDR模板作为双链DNA gBlock订购,并克隆到pSmart载体中(例如,pSMART-HCKan, CloneSmart Blunt Cloning kit)。候选gRNA作为单链DNA寡核苷酸订购,退火并克隆到pX330家族的载体中(Cong等人,2013年)。补充文件中提供了基本的克隆协议。
设计和克隆后,使用NucleoBond Xtra Midi Plus EF试剂盒对编码HDR和gRNA模板的质粒DNA进行中量提取,并使用标准引物进行测序。A260:A280比率应至少为1.8。
我们已经开发了能够在大鼠IgG2a杂交瘤中修改HC的质粒,用新的同种型替换整个恒定区,以及在铰链处剪切并插入任何感兴趣的标签(Fab’片段)(van der Schoot等人,2019年),以及修改小鼠杂交瘤的mIgG1 CH1(如图2-6所示并在视频中记录)和Cκ kappa LC(Le Gall等人,2021年)。我们发布的质粒可在Addgene上找到或通过请求获得。使用标准的分子克隆技术和专用的限制性位点,可以轻松地在HAs之间交换插入物。表1中报告了gRNAs,表5中报告了HAs(注释)。
C. 杂交瘤细胞的培养及转染前抗生素的滴定
在抗生素压力下进行HDR重组成功的细胞的筛选。在进行该程序之前确保亲本细胞系的质量至关重要(参见注释)。目标杂交瘤应在实验开始前进行抗生素(博来霉素/嘌呤霉素)敏感性测试。
- 在完整的培养基中培养目标杂交瘤细胞,无论是自建的还是从美国典型培养物保藏中心获得的,确保细胞保持高活力(> 90%)。
- 当细胞汇合时,将1 × 10^6细胞/孔接种到6孔板中,加入3 mL的培养基,并补充0、0.5、1、2、4、8、10或20 μg/mL的适当抗生素。
- 在37°C下培养细胞3-7天,具体时间取决于选择的抗生素(嘌呤霉素为3天,博来霉素为7天)。
- 使用台盼蓝确定在培养基中没有存活细胞的最小浓度,并使用确定的浓度进行筛选程序(图7A-B)。
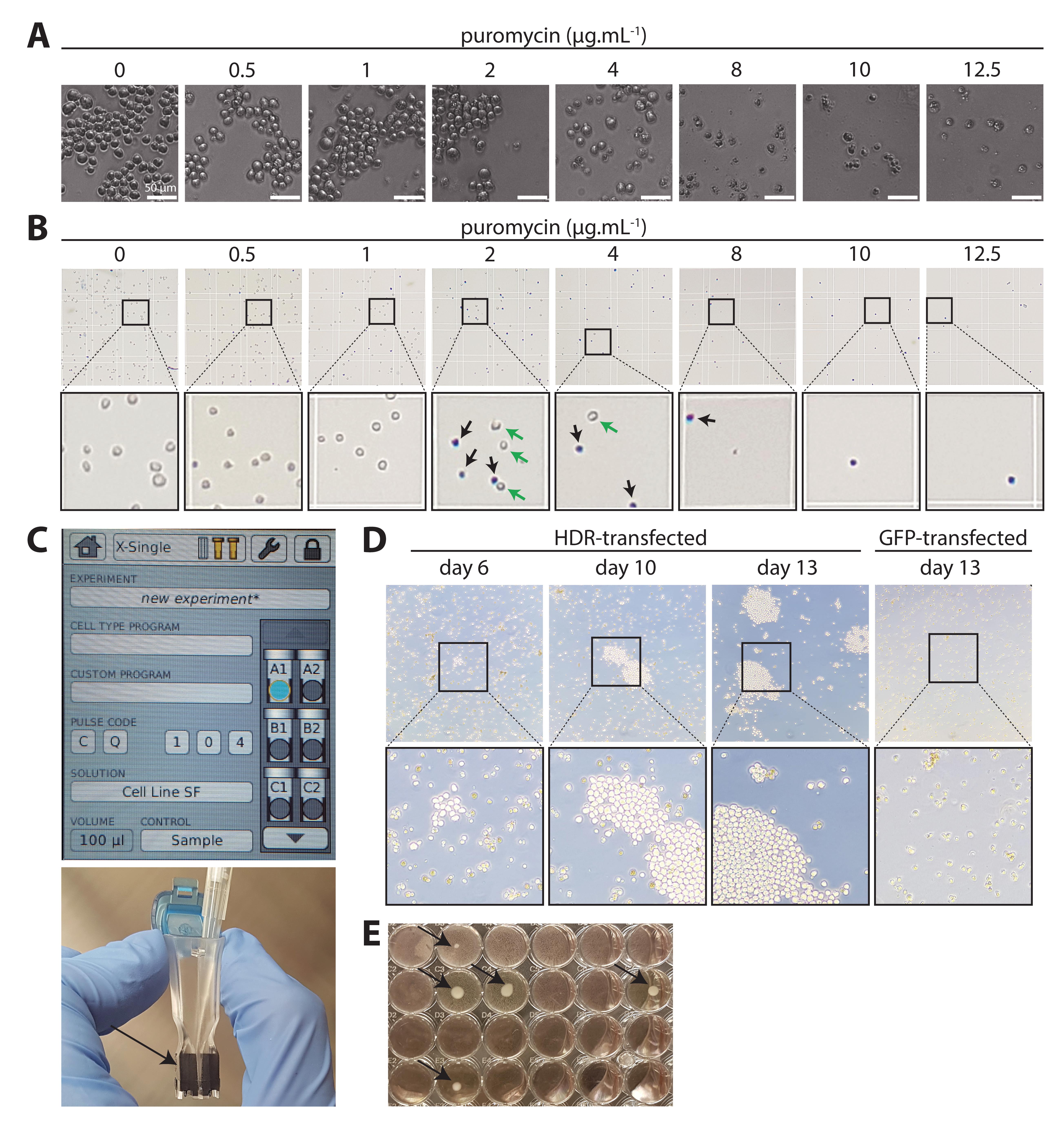
[^图 7. 抗生素滴定、转染和耐药细胞扩增。]: A. 对野生型(WT)抗人CD20 [NKI.B20/1] 进行普鲁霉素滴定。普鲁霉素的连续稀释浓度为0–12.5 μg/mL。48小时后,处理组细胞开始死亡。健康的杂交瘤细胞呈圆形且规则,并成簇生长。死亡或濒死的细胞失去圆形,变得不规则。在0.5 μg/mL时可观察到死亡细胞,且在4 μg/mL时仍有存活细胞。从8 μg/mL开始,细胞明显死亡。经过训练的眼睛可以轻松识别细胞的存活情况。B. 为了量化存活率,使用台盼蓝染色区分活细胞(白色)和死细胞(蓝色)。这里我们选择8 μg/mL普鲁霉素进行实验。C. 使用AMAXA电转染杯和程序(细胞系SF,CQ-104)。小心地将含有细胞和DNA的悬液用移液管吸入电极之间(箭头),避免产生气泡。D. 经过筛选后,HDR转染的培养皿中开始出现小簇生长,大部分细胞死亡或濒死(第6天)。这些耐药细胞成功进行了HDR。继续培养,直至更大的细胞簇出现(第10天),并逐渐长满培养皿(第13天)。相比之下,绿色荧光蛋白(GFP)转染的杂交瘤细胞保持死亡状态,未见细胞簇(第13天,右图)。当培养皿长满后,进行限稀释。E. 经过限稀释后,单克隆细胞系可以在各自井底的底部轻易识别(箭头)。扩增并鉴定这些细胞系。
D. 杂交瘤细胞的转染
本节描述了使用gRNA/Cas9 + HDR模板或绿色荧光蛋白(GFP)作为模拟转染对照,对1 × 106野生型杂交瘤细胞进行电转染的过程。将细胞长时间悬浮在SF转染介质中(来自SF Cell Line 4D-NucleofectorTM X试剂盒)是有害的:需快速、有序地进行转染,并依次进行。
- 在第0天,使用高存活率(>90%)的野生型杂交瘤细胞进行电转染。
- 向6孔板中加入3 mL培养液,并在37 °C下孵育。用50 mL Falcon预先加热培养液。
- 从所需细胞系中离心沉淀杂交瘤细胞(1,500 rpm,5分钟,4 °C)。
- 用适量的洗涤缓冲液(目标是1 × 106–2 × 106细胞/mL)重新悬浮细胞沉淀。
- 使用台盼蓝计数细胞,并将1 × 106细胞转移到1.5 mL Eppendorf管中。
- 在台式离心机中离心细胞悬浮液(1,500 × g,5分钟,室温)。
- 在此期间,准备两个或所需数量的DNA混合物,在0.2 mL Eppendorf管中,通过用移液管吸取每个条件下1 μg的Cas9/gRNA + 1 μg的HDR模板,以及2 μg的pMAX-GFP作为模拟转染对照(DNA混合管)。
- 使用参数:程序CQ-104,细胞系SF,准备4D-Nucleofector。
- 小心吸走上清液。
- 向细胞沉淀中加入100 μL SF转染介质,并在转移到含有HDR模板+ gRNA或pMAX-GFP的DNA混合管之前重新悬浮细胞。
- 重新悬浮,并小心地将悬液转移到Lonza电转染试剂盒提供的100 μL电转染杯底部(图7C)。轻轻敲击电转染杯以确保样本覆盖电极和电转染杯底部,并检查是否有气泡。
- 关闭盖子,将电转染杯插入AMAXA电转染器中。按显示屏上的开始按钮,使用参数:程序CQ-104,细胞系SF(图7C)进行电转染。
- 小心地向电转染杯中加入1 mL预热的培养基,并将细胞立即转移到含有预热培养基的6孔板中(孔中总体积现在约为4 mL)。避免反复吸取细胞。
- 用培养基冲洗电转染杯,以确保所有细胞都已转移。
- 进行下一次转染(从步骤8开始重复)。
- 将6孔板在37 °C的湿化CO2培养箱中孵育。
E. 工程杂交瘤细胞的筛选及单克隆细胞系的生成
本节描述了筛选成功工程化的杂交瘤细胞以识别HDR编辑克隆的过程。这是通过从转染后第3天起对HDR-和GFP-转染的杂交瘤细胞施加抗生素选择压力来完成的。耐药细胞生长至融合,并可生成单克隆细胞系。
转染细胞在无选择压力的培养液中培养两天,以允许电转染的细胞生长。在第1天,通过目测检查转染细胞的存活率,并使用荧光显微镜检查转染对照细胞中的GFP表达。小心处理细胞以保持其存活率。
在第3天,刮取细胞或重新悬浮直至细胞脱离,并将所有4 mL的细胞悬液转移到含有5 mL预热的1倍选择培养基和两倍预定浓度的适当选择药物(2×选择培养基)的10 cm培养皿中。用1 mL培养液冲洗井,收集所有细胞并转移到培养皿中。转移细胞后,应使用培养液调整培养皿中的最终体积至10 mL,以确保细胞处于1×选择培养基中。
从第5天开始,每周两次传代转染细胞。具体操作为:刮取细胞,转移到15 mL Falcon管中。在10 cm培养皿中放置5 mL新的预热的1×选择培养基。离心(1,500 rpm,5分钟,室温)并弃去上清液。在2 mL预热的1×选择培养基中重新悬浮细胞沉淀,并将其悬液转移回培养皿。用3 mL 1×选择培养基冲洗15 mL Falcon管,并将其转移回培养皿。
根据抗生素的不同,到第5-10天,HDR/gRNA转染的培养皿中应在大多数死亡细胞中形成小簇,而GFP转染的细胞全部死亡(图7D)。如果没有观察到细胞死亡,应调整抗生素浓度(见注释)。如果在抗生素压力下没有活细胞生长,请参阅列出的故障排除解决方案(见注释)。
继续传代细胞,直到这些簇生长达到更高的融合度和存活率(图7D,>90%,通常在第15-20天左右)。当HDR转染的培养皿长满时,GFP培养皿中的细胞仍应全部死亡。此时,丢弃GFP转染的培养皿,并继续进行HDR转染细胞的限稀释。在此步骤之后,不再需要抗生素压力。如果有些簇生长非常缓慢或GFP转染的培养皿中仍有存活的细胞,最好在鉴定单克隆细胞系后继续在培养基中保留所选抗生素。
在五个圆底96孔板中预先加入100 μL/孔的培养液,并在37 °C下孵育。
刮取融合的HDR转染细胞并离心细胞悬液(1,500 rpm,5分钟,室温)。保留1 mL上清液样本,并丢弃其余部分。
在5 mL培养液中重新悬浮细胞并使用台盼蓝计数。在培养液中分两步稀释细胞,以获得浓度为3细胞/mL的50 mL悬液。
[示例:初始悬液浓度为1.5 × 106细胞/mL → 稀释因子:500,000(1.5 × 106/3)。首先进行500倍稀释,100 μL细胞悬液在50 mL培养液(3,000细胞/mL)中。然后取50 μL细胞悬液(3,000细胞/mL)在50 mL培养液中稀释1,000倍(浓度为3细胞/mL)。]
在圆底96孔板中每孔加入100 μL悬液。最终浓度为0.3细胞/孔在200 μL或33细胞/板。
从原始细胞悬液中保留约500,000个细胞用于基因组DNA(gDNA)提取(使用ISOLATE II Genomic DNA提取试剂盒)。离心并遵循制造商的说明。
按照标准冷冻保存程序冷冻保存大量群体的剩余细胞。
在单细胞克隆(SCC)生长的同时,应立即对大量群体及其上清液进行表征,以确定HDR模板的整合。为此,请遵循数据分析部分1-3的内容。
F. 修改后杂交瘤细胞的扩增
- 在37 °C下孵育培养板10-15天,期间不更换培养基。
- 当在皿底可见SCC(单细胞克隆)(图7E)时,将它们转移到新的平底96孔板中,并加入100 μL新的培养基。
- 当细胞融合时,收集并冷冻上清液,并将克隆扩展到24孔板,然后是T75烧瓶。
- 收集约500,000个细胞用于gDNA提取,并冷冻保存扩大的克隆。
数据分析
选择克隆的表征(来自大量群体和/或单细胞克隆群体)
验证细胞系是否产生正确的抗体(片段)至关重要。首先在大批量群体上进行筛选方法,以确保程序的顺利进行,然后在单细胞克隆上进行。我们通常结合三种或更多方法来确保插入片段的整合和正确的蛋白生产:在基因、蛋白质和功能层面上。下面提供了几个不同方法的例子以供说明。在功能表征之前,这个直接的协议不需要进行统计分析。如果耐药细胞没有产生修改后的抗体,请参阅列出的故障排除解决方案(见注释)。
为了说明这一部分,我们展示了使用此技术修改的三个不同细胞线的全面表征:
- MIH5是一种产生单克隆抗体的鼠IgG2a, λ杂交瘤,这些抗体与小鼠PD-L1结合,这是一种由癌细胞和髓系细胞表达的蛋白。PD-L1是基于mAb(抗体药物)治疗的癌症(检查点抑制剂)的治疗靶点。我们工程化了MIH5细胞系,以产生具有鼠IgG2a(m2a)和鼠IgG2a L234A/L235A/N297A(m2a(silent))同种型的嵌合抗PD-L1抗体(图8A)(van der Schoot等人,2019年)。
- NLDC-145是一种产生单克隆抗体的鼠IgG2a, κ杂交瘤,这些抗体与小鼠DEC-205结合,DEC-205是一种由能够交叉呈递抗原的树突细胞亚群表达的C型凝集素。由于其表达受限,DEC-205是开发靶向疫苗的有前景的靶点,这些疫苗能有效地将抗原递送到能够最佳激发T细胞反应的受限细胞群体。我们工程化了NLDC-145以产生截短的抗体(Fab’片段),这些抗体带有来自模型抗原卵清蛋白(OVA)的CD8 T或CD4 T细胞表位(分别为Fab’OTI和Fab’OTII)(图8B)(Fennemann等人未发表的数据,2021年)。
- NKI.B20/1是一种产生单克隆抗体的鼠IgG1,κ杂交瘤,这些抗体与人CD20结合。CD20是Rituximab的靶点,Rituximab是一种用于治疗B细胞淋巴瘤、类风湿性关节炎或狼疮的B细胞耗竭mAb。我们生成了一种NKI.B20/1细胞系,该细胞系产生一个Fab’片段,在HC和LC上带有两个正交的位点选择性修饰位点(图8C):HC上的LAETGG-His标签和LC上的LPESGG-Myc标签。这个构建体可以用来生成带有两个不同有效载荷的均质抗体-药物偶联物(Le Gall等人,2021年)。
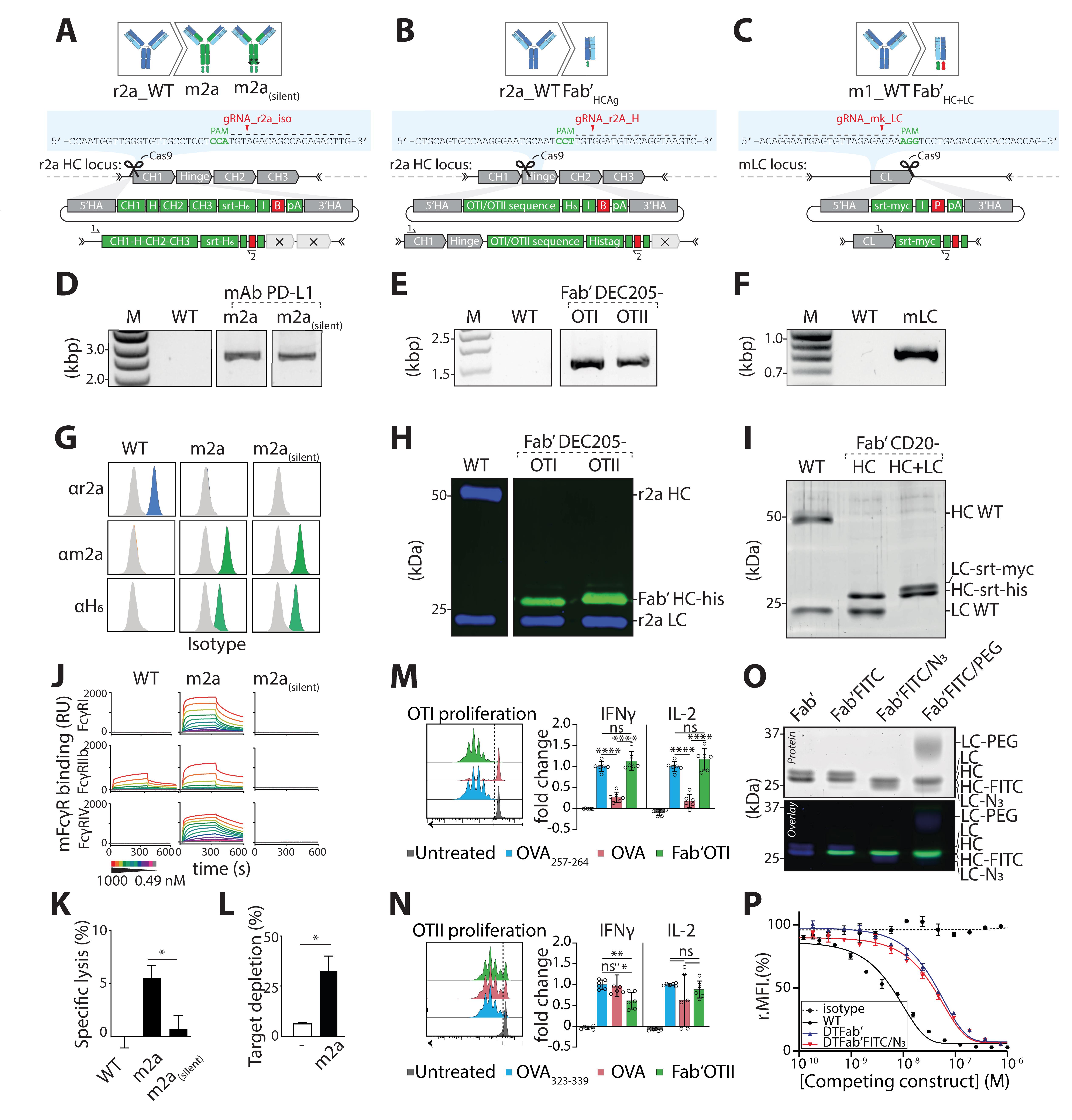
[^图 8. 在基因、蛋白质和功能层面上对三种工程细胞系进行验证]: 。A. 用于将大鼠IgG2a杂交瘤转变为小鼠IgG2a的HDR模板和gRNA的等位基因转换。B. HDR模板结构和用于将大鼠IgG2a杂交瘤转变为Fab’抗原(Fab’Ag)的gRNA。此处展示的抗原是OTI和OTII肽。C. 用于编辑小鼠IgG1杂交瘤LC的HDR模板和gRNA。在此策略中,HC和LC都经过编辑,以形成一个带有两个选择性修饰位点的Fab’片段(此处展示LC编辑)。D. 使用位置特异性(1)和HDR特异性(2)引物集,对从m2a和m2a(silent)抗性克隆中提取的gDNA进行PCR扩增。E. 使用位置特异性(1)和HDR特异性(2)引物集,对从Fab’OTI和Fab’OTII抗性克隆中提取的gDNA进行PCR扩增。F. 使用位置特异性(1)和HDR特异性(2)引物集,对从mκ抗性克隆中提取的gDNA进行PCR扩增。G. 在选择每种等位基因的单克隆杂交瘤后,将上清液与表达PD-L1的目标细胞(CT26)孵育,并用抗r2a、抗m2a或抗His标签染色。显示的图表明上清液仅含有带C端His标签的工程等位基因变体,而原始的大鼠IgG2a单抗不存在。H. Western blot Fab’ -OTI和-OTII,染色His标签(绿色)和大鼠IgG2a(蓝色)。使用NLDC-145抗体作为阴性对照(WT)。I. SDS-PAGE凝胶内荧光(Sypro染色)显示WT mAb CD20、Fab’HC和Fab’HC+LC蛋白的分子量在编辑HC和LC后发生改变。J. 代表性传感图显示MIH5 WT和MIH5工程单抗(WT r2a、m2a、m2asilent)与固定的小鼠FcγR(FcγRI、FcγRIIb和FcγRIV)在递增浓度(0.49–1000 nM)下的相互作用。与FcγR的结合以共振单位(RU)表示。K. 体外抗体依赖性细胞毒性(ADCC)试验比较MIH5等位基因变体的效应功能。将MC38细胞用51Cr标记,用m2a或m2a(silent) MIH5等位基因变体进行调理,然后暴露于C57BL/6小鼠的全血中4小时。通过测量51Cr释放(n = 3,平均值±SEM,* p < 0.05)来量化特异性裂解,并显示m2a可诱导PD-L1表达细胞的特异性裂解,而m2a(silent)在体外无活性。L. 体内耗竭试验。使用Violet和Red示踪染料标记的脾脏B细胞作为体内耗竭的目标细胞,并用m2a(silent)变体或m2a进行调理。随后,将B细胞以1:1的比例(m2a:m2a(silent)或m2a(silent):m2a(silent))混合并静脉注射到C57BL/6小鼠中。24小时后,分离脾脏,并通过流式细胞术测定荧光标记细胞群之间的比例,以量化目标细胞的等位基因特异性耗竭,显示m2a和m2a(silent)在体内均具有功能(n = 3,平均值±SEM,* p < 0.05)。M. 使用Flt3L和OP9-DL-1饲养层(Kirkling等人,2018年)从骨髓生成表达DEC-205的小鼠树突状细胞。在第8天,使用CD11c微珠分离CD11c+细胞,并与1 μM Fab’OTI、5 nM OVA257-263(SIINFEKL,阳性对照)或10 μM OVA孵育2小时。洗涤后,将细胞暴露于新鲜分离的荧光标记OTI CD8 T细胞中,并补充0.3 μg/mL脂多糖(LPS)。72小时后,通过流式细胞术分析细胞增殖(细胞染料稀释)和活化标志物(未显示),并通过ELISA分析上清液中促炎IFNγ和IL-2细胞因子的产生。Fab’OTI处理的树突状细胞诱导CD8 T细胞增殖和活化,表明它被DEC-205表达的树突状细胞摄取并加工,并且CD8 T细胞表位在I类主要组织相容性复合体(MHC)上呈现(n = 3,平均值±SD,ns:不显著,**** p < 0.0001,单因素方差分析后Tukey多重检验)。N. 第8天表达DEC-205的小鼠树突状细胞与1 μM Fab’OTII、1 μM OVA323-339(ISQAVHAAHAEINEAGR,阳性对照)或10 μM OVA孵育2小时。洗涤后,将细胞暴露于新鲜分离的荧光标记OTII CD4 T细胞中,并补充0.3 μg/mL LPS。72小时后,通过流式细胞术分析细胞增殖和活化标志物表达(未显示),并通过ELISA分析上清液中促炎IFNγ和IL-2细胞因子的产生。Fab’OTII处理的树突状细胞诱导CD4 T细胞增殖和活化,表明它被DEC-205表达的树突状细胞摄取并加工,并且CD4 T细胞表位在II类MHC上呈现(n = 3,平均值±SD,ns:不显著,* p < 0.05,** p < 0.01,单因素方差分析后Tukey多重检验)。O. SDS-PAGE分析Fab′CD20顺序特异性标记H-GGG-C-K(FITC)-NH2在HC上和H-GGG-K(N3)-NH2在LC上,确认在工程Fab′上引入了两个不同的货物,并且引入的基序功能正常。P. 抗原结合竞争试验,每个工程蛋白对mAbNKI.B20/1-AF647的结合亲和力表明,经过CRISPR/Cas9编辑和顺序双重特异性标记后,蛋白质没有失去对它们目标的结合亲和力。B: 阳光霉素抗性基因,用于在细胞培养中筛选成功转化的细胞。FITC: 荧光素异硫氰酸酯,一种常用的荧光标记物。 H: 铰链区,连接抗体轻链和重链的结构域。 H6: 六聚组氨酸标签(HHHHHH),用于蛋白质纯化和检测。 HA: 同源臂,在基因编辑中用于引导DNA修复。 HC: 重链,抗体的一部分,负责与抗原结合。 HDR: 同源定向修复,一种基因编辑技术,利用DNA的天然修复机制插入或替换DNA序列。 I: 内部核糖体进入位点(IRES),一种允许在翻译水平上调控基因表达的结构。 IFNγ: 干扰素gamma,一种免疫调节蛋白,具有抗病毒和抗肿瘤活性。 IL-2: 白细胞介素-2,一种免疫调节蛋白,促进T细胞增殖和分化。 LC: 轻链,抗体的一部分,与重链一起构成抗体的可变区。 myc: myc标签(EQKLISEEDL),一种常用的蛋白质标签,用于检测和纯化。 OVA: 卵白蛋白,常用于免疫学研究中的抗原。 P: 阳光霉素抗性基因,用于在细胞培养中筛选成功转化的细胞。 pA: 聚A尾,存在于真核生物mRNA末端的序列,有助于mRNA的稳定和翻译。 PAM: 间隔相邻基序,CRISPR/Cas9系统中指导RNA识别目标DNA序列的关键序列。 PD-L1: 程序性死亡配体1,一种免疫检查点分子,在肿瘤免疫逃逸中起重要作用。 PEG: 聚乙二醇,一种多功能聚合物,常用于蛋白质化学和药物输送。 srt: sortag motif,一种用于蛋白质修饰的短肽序列。 WT: 野生型,指未经基因改造的自然基因型。
PCR扩增目标位点以确定HDR整合
应该对细胞团和SCC(单细胞克隆)群体进行PCR筛选。为此,使用从约500,000个细胞中分离的gDNA,并使用仅与插入序列杂交的引物以及与插入位点外HA区域附近杂交的位置特异性引物扩增目标位点。使用约100 ng的gDNA,使用标准PCR程序(在补充文件中提供)扩增目标位点。表2提供了用于扩增插入我们的HDR模板后的鼠IgG1重链、鼠κ轻链和大小鼠IgG2a CH1重链的引物序列。Table 2. Locus-specific and HDR-specific for PCR amplification of target region
Primer Target 5′-3′ Sequence r2a_iso_fw rat IgG2a upstream of CH1 (locus-specific) GGCGACCTGTAACAACTTGG r2a_CH1_fw rat IgG2a CH1 exon (locus-specific) TGTAGGAGCTTGGGTCCAGA m1_CH1_fw mouse IgG1 CH1 (locus-specific) GTGCCGACTTCAATGTGCTT mκ_LC_fw mouse kappa light chain (locus-specific) GTGCTTGTGTTCAGACTCCC blast_rv blasticidin resistance gene (HDR-specific) ATACATTGACACCAGTGAAGATGC ires_rv IRES (HDR-specific) GGCTTCGGCCAGTAACGTTA polyA_rv poly A tail (HDR-specific) CATAGAGCCCACCGCATCCC a. 将PCR产物在1%(w/v)的琼脂糖凝胶上运行,并使用Nancy-520(1:15,000)可视化DNA。
b. 插入物在目标区域的整合通过存在预期的工程MIH5 [r2a>m2a和r2a>m2a(silent) (r2a_iso_fw + blast_rv, Figure 8D)], NLDC-145 (r2a>Fab’OTI和r2a>Fab’OTII (r2a_HC_fw + blast_rv, Figure 8E)), 和NKI.B20/1 [mκ> mκ-srt-myc (mκ_LC_fw + ires_rv, Figure 8F)] 细胞系的单个带得到确认。
c. 应在正确的高度切割正确的带并进行凝胶纯化,以进行Sanger测序并确认正确的整合。杂交瘤上清液的流式细胞术分析
可以进行流式细胞术筛选,以研究例如His标签/Myc标签、重链、轻链或某种等位基因的表达。
对于初步筛选,将50 μL的细胞团或SCC的上清液与50,000个目标表达细胞在4°C下孵育20分钟。在PBA洗涤两次后,进行二次染色,使用表3中指示的抗体来染上清液产生的抗体(等位基因、标签等)。Table 3. Secondary antibodies used to analyze hybridoma production by flow cytometry
Target Clone Fluorophore Company Cat. number Dilution 6×His-tag J095G46 PE Biolegend 362603 25 rat IgG2a HC MRG2a-83 PE Biolegend 407507 400 mouse IgG2a m2a-15F8 PE eBioscience 12-4817-82 500 对于旨在选择高产量克隆的第二次筛选,使用类似的工作流程,但包括积极克隆的上清液的连续稀释。建议带有一个已知浓度的亲本抗体的稀释液,以计算样本中的抗体量。
我们修改了产生r2a的MIH5细胞系,使其产生m2a和m2a(silent)等位基因,并将积极克隆的未稀释上清液与表达PD-L1的细胞(Figure 8G)孵育。流式细胞术分析显示,最初细胞系产生原始等位基因,但未选择克隆,并产生带有His标签的m2a等位基因,证实了正确的整合。Western blot分析杂交瘤上清液
流式细胞术筛选可以用来研究例如His标签/Myc标签、重链、轻链或某种等位基因的表达,而不需要大量的目标表达细胞。必须在选定的克隆上单独确认与目标的结合。
我们通常首先使用点印迹进行筛选。为此,取每个单细胞克隆上清液的3 μL,并在硝酸纤维素膜上点印,使用96孔板作为指南。让硝酸纤维素膜干燥30分钟,并进行阻断步骤和一般Western blot协议的二次染色,使用表4中指示的抗体来染上清液产生的抗体(等位基因、标签等)。
对于第二次筛选,使用积极克隆的上清液的稀释液来确认正确蛋白的生产,使用传统的Western blot。在这里,我们分析了NLDC-145工程细胞系产生的Fab’OTI和Fab’OTII上清液,使用抗rIgG2a(H+L)和抗His标签(Figure 8H)。结果显示,产生了一个带有His标签的25 kDa的重链,高于野生型(WT)50 kDa的重链,确认了HDR插入。
另一种方法是SDS-PAGE分析可以确认正确产品的生产(Figure 8I)。在这里,我们依次将NKI.B20/1亲本细胞系修改为一个产生带有两个位点特异性修饰位点的Fab’片段的细胞系,分别在其重链和轻链上。在第一次和第二次修改后,我们分别检测到重链从50 kDa降至25 kDa(WT重链 > Fab’-srt-his重链),以及轻链的向上移动(WT轻链 > LC-srt-myc轻链)(Figure 8I)。
Table 4. Antibodies used to characterize presence of the right insertTarget Clone Fluorophore Company Cat. number Dilution rabbit anti 6×His-tag RM146 unconjugated Abcam AB14923 1,000 goat anti-rabbit IgG (H + L) polyclonal IRD800 LI-COR 926-32211 5,000 goat anti-rat IgG (H + L) polyclonal AF680 Thermo Fisher A-21096 5,000 确认生产功能性蛋白
确认选定克隆能产生功能性抗体对于验证选定的细胞系至关重要。a. 高水平的修改抗体生产
基于基因组筛选和蛋白质水平确认,可以根据其生产能力和生长速率选择一个克隆,用于生产高水平的修改抗体。为此,按照制造商的说明,将选定的克隆扩大到5层培养瓶或CELLineCL 1000生物反应器。b. 修改抗体的分离
分离方法取决于插入物的性质。HDR模板可以设计包括纯化标签,如His标签或Myc标签,对于这些标签,亲和树脂是可用的(例如,Ni-NTA琼脂糖)。不包含此类标签的抗体可以使用例如蛋白G树脂进行分离,在这种情况下,培养基需要无血清(CD杂交瘤培养基)。c. 功能测定
确认抗体功能所需的测定通常取决于修饰的性质、目标抗原和预期用途。在这里,我们提供了我们进行的功能测定的例子,以验证所呈现的细胞系。这些结果表明,在免疫球蛋白位点中进行的CRISPR/Cas9敲入保留了抗体的特异性,并且插入物是功能性的。我们生成了产生m2a和m2a(沉默)等位基因的MIH5细胞系。为了确认等位基因的功能,我们进行了表面等离子共振分析,以评估纯化的mAbs在固定的小鼠可溶性片段结晶γ受体(FcyR)上的结合能力(图8J)。正如预期的那样,m2a抗体与小鼠FcyR结合,并且与m2a(沉默)中引入的突变结合被阻断。此外,我们进行了不同等位基因(图8K)包被的PD-L1表达癌细胞的原发性细胞毒性实验。与C57BL6/J小鼠的全血共培养显示,m2a能够特异性裂解靶细胞,而r2a(WT)和m2a(沉默)则没有活性。类似地,在体内杀伤实验中,与m2a或m2a(沉默)共培养的脾细胞显示出对m2a处理细胞的特定杀伤作用(图8L)。这些结果共同验证了新型嵌合抗体的功能(van der Schoot等人,2019年)。
工程化的NLDC-145细胞系产生Fab’OTI和Fab’OTII,这些蛋白可以特异性地将抗原递送到表达DEC-205的树突状细胞(DCs)。OTI和OTII抗原分别被CD8 OTI和CD4 OTII T细胞识别,这些T细胞在DCs呈递的MHC分子上。我们用构建体、短肽(阳性对照,OVA257-264或OVA323-339)或OVA蛋白孵育DEC-205表达的初级小鼠树突状细胞2小时,然后将DCs暴露于抗原特异性CD8或CD4 T细胞(图8M-N),这些细胞用CTV标记。CD8(图8M)和CD4(图8N)T细胞在识别其相应抗原后增殖并产生促炎细胞因子,这些细胞因子通过流式细胞术测定其增殖(CTV稀释),通过表达激活标志物,并通过ELISA检测细胞因子产生(IL-2,IFNγ)。当DCs暴露于短肽和Fab’抗原融合蛋白时,CD8和CD4 T细胞增殖并产生促炎细胞因子。这证实了Fab’抗原融合蛋白保留被DCs处理和呈递的能力,并且强烈诱导T细胞激活(Fennemann等人未发表的数据)。
最后,我们生成了一个产生带有正交位点特异性修饰位点的Fab’片段的NKI.B20/1细胞系,这些位点位于重链和轻链的C末端(Le Gall等人,2021年)。为了确认正交位点的功能,我们依次修改重链以引入荧光素异硫氰酸酯,修改轻链以引入叠氮基团,这在荧光SDS-PAGE上得到确认(图8O)。最后,我们通过在表达CD20的目标细胞上进行竞争结合实验来确认位点特异性修饰不会改变与靶标的结合(图8P)。
Notes:
我们已经成功地将此协议应用于各种杂交瘤细胞系。我们建议操作者在进行遗传工程之前,对目标细胞系的生长速率、产量水平和存活率有一个良好的了解。我们发现以下细节可以帮助完善程序:
确保亲本细胞系的质量
杂交瘤可能会随着时间的推移而失去生产能力或存活率低,这严重损害了程序的效率。为了解决这个问题,我们建议操作者通过执行限制稀释(如在E节中描述)来亚克隆亲本线,并量化SCC的存活率和WT抗体产量,以选择一个新的单克隆线。这确保了选择高度存活的亲本克隆和高产量的细胞。在亲本细胞系上进行亚克隆还可以确认SCC能够生长。如果亲本线的存活率没有改善,调整培养条件,例如通过去除β-ME(确保抗体生产不受影响)或在培养基中增加FBS浓度,以800 rpm而不是1,500 rpm的转速离心5分钟,或使用密度梯度如Lymphoprep去除死细胞。确认亲本线对抗生素压力的敏感性
通常,杂交瘤细胞系对选择性抗生素没有抵抗力,我们不需要对模拟转染的杂交瘤进行抗生素滴定。对于每个细胞系可能不是这样,并且在某些情况下,需要在模拟转染的杂交瘤上进行滴定。如果是这种情况,请按照D节模拟转染细胞系,然后按照C节中的指示进行滴定。在程序过程中调整选择药物的浓度
细胞死亡通常从第2-3天开始。对于嘌呤霉素,细胞应在第7天死亡,对于阳光霉素,应在第10天死亡。在抗生素压力下,我们通常观察到GFP和HDR转染板的细胞大量死亡。如果情况并非如此,选择药物的浓度太低。即使经过滴定,也可能发生这种情况,操作者应增加浓度。如果转染细胞在抗生素压力下不能生长
如果转染细胞不能承受抗生素压力,可以尝试以下解决方案。在转染前检查杂交瘤的存活率并进行GFP转染效率。序列目标区域以确保没有可能影响gRNA/HDR效率的突变。进行T7实验以确定gRNA切割目标位点的能力,并在必要时尝试其他gRNAs。测试支原体污染。通过饥饿(低/无FBS)同步转染前的细胞。加入非同源末端连接抑制剂,如nocodazole,以促进HDR。如果在限制稀释后SCCs不能生长
虽然我们从未遇到过这个问题,但一些细胞系在限制稀释后可能无法生长。为了解决这个问题,可以在培养基中加倍FBS浓度来拯救单细胞。此外,使用条件培养基进行限制稀释可以帮助单细胞存活。为了制备条件培养基,在10厘米的培养皿中接种约1 × 106个亲本细胞。根据生长速率,24小时或48小时后,离心上清液并通过0.45微米过滤器。如果抗性细胞不产生(仅)修改的抗体
在抗性克隆中,我们通常发现有很大比例的克隆正确编辑并产生大量抗体(50%–90%)。我们发现偶尔有SCC能够抵抗抗生素压力并显示出插入物的正确整合,但产生WT抗体,我们认为这是等位基因排除的例外情况。通常,其他SCC会产生正确的蛋白,应该选择这些克隆。
然而,如果所有在抗生素压力下存活的SCC通过PCR在目标区域显示出插入物的整合,但不分泌修改的抗体,则HDR模板可能需要调整。例如,由于替代剪接,SCC可以抵抗抗生素压力但产生修改和WT抗体,或者只产生WT抗体。为了解决这个问题,删除3’端接头序列中插入下游的exon的剪接供体序列,并在合成exon上游插入一个合成的剪接供体序列(如GCTAGCGATCGCAGGCGCAATCTTCGCATTTCTTTTTTCCAG)。特别是如果敲入上游的exon,请确保在剪接后插入序列与变异性别的序列对齐。表5. 本说明中使用的同源臂。粗体:用于在成功HDR重组后失活Cas9的突变PAM序列。另一种选择是在供体序列中完全切除(例如,Fab r2a_HC)。下划线:原始剪接供体位点位于rIgG2a CH1 exon上游:在HDR模板中删除或突变以防止与原始等位基因的变异性别区域发生替代剪接。
| Homology arms | 5′-3′ Sequence |
|---|---|
| 5′ HA r2a_iso (rIgG2a WT > mIgG2a) | AGAAAGATCTGAGTAGAACCAAGGTAAAAAGTGTGGGTAAAAACACATGTTCACAGGCCTGGCTGACATGATGCTGGGCACGTATGGAGGCAAAGTCAAGAGGGCAGTGTAAGGGCCAGAAGTGAATCCTGACCCAAGAATAGAGAGTGCTAAACCTACGTAGATCGAAGCCAACTAAAAAGACAAGCTACAAAACGAAGCTAAGGCCAGAGATCTTGGACTGTGAAGAGTTCAGAGAACCTAGGATCAGGAACCATTAGTAACAGGCCAAGGAAGATAGAAGCTGCCTAGGACTTGGCAAGAGCCAACATGGTTGGACTGGAAAAGAAAGGAGGAGACAGAAGACAGGAGAGATGTGCCAACTTGATTTTGGGCTTCACTGTTGTCCATACTGTGTGCAGCCATATGGCCCACAGATAACAGGTTTAGCCGAGGAACACAGATACCCACATTGGACAATGGTGGGGGAACACAGATACCCATACTACAGGGCTCTTTAGGGCATTTCCTGAAAGTGTACTAGGAGTGGGACTGGGCTCAAAGGGATTAGGTGTGATCTGGCCTGGTGAGGCTGACATTGGCAAGCCCAATGGTTGGGTGTTGCCTCCTCCATGT |
| 3′ HA r2a_iso (rIgG2a WT > mIgG2a) | TGTACAACTTGGGGAGGGTACAAAATGGAGGACTTGTAGGAGCTTGGGTCCAGACCTGTCAGACAAAATGATCACGCATACTTATTCTTGTAGCTGAAACAACAGCCCCATCTGTCTATCCACTGGCTCCTGGAACTGCTCTCAAAAGTAACTCCATGGTGACCCTGGGATGCCTGGTCAAGGGCTATTTCCCTGAGCCAGTCACCGTGACCTGGAACTCTGGAGCCCTGTCCAGCGGTGTGCACACCTTCCCAGCTGTCCTGCAGTCTGGACTCTACACTCTCACCAGCTCAGTGACTGTACCCTCCAGCACCTGGTCCAGCCAGGCCGTCACCTGCAACGTAGCCCACCCGGCCAGCAGCACCAAGGTGGACAAGAAAATTGGTGAGAGAACAACCAGGGGATGAGGGGCTCACTAGAGGTGAGGATAAGGCATTAGATTGCCTACACCAACCAGGGTGGGCAGACATCACCAGGGAGGGGGCCTCAGCCCAGGAGACCAAAAATTCTCCTTTGTCTCCCTTCTGGAGATTTCTATGTCCTTTACACCCATTTATTAATATTCT |
| 5′ HA r2a_HC (rIgG2a WT > Fab’ fragment) | CCTGGAACTCTGGAGCCCTGTCCAGCGGTGTGCACACCTTCCCAGCTGTCCTGCAGTCTGGACTCTACACTCTCACCAGCTCAGTGACTGTACCCTCCAGCACCTGGTCCAGCCAGGCCGTCACCTGCAACGTAGCCCACCCGGCCAGCAGCACCAAGGTGGACAAGAAAATTGGTGAGAGAACAACCAGGGGATGAGGGGCTCACTAGAGGTGAGGATAAGGCATTAGATTGCCTACACCAACCAGGGTGGGCAGACATCACCAGGGAGGGGGCCTCAGCCCAGGAGACCAAAAATTCTCCTTTGTCTCCCTTCTGGAGATTTCTATGTCCTTTACACCCATTTATTAATATTCTGGGTAAGATGCCCTTGCATCATGACATACAGAGGCAGACTAGAGTATCAACCTGCAAAAGGTCATACCCAGGAAGAGCCTGCCATGATCCCACACCAGAACCAACCTGGGGCCTTCTCACCTATAGACCATACTAACACACAGCCTTCTCTCTGCA |
| 3′ HA r2a_HC (rIgG2a WT > Fab’ fragment) | GGTAAGTCACTAGGACTATTACTCCAGCCCCAGATTCAAAAAATATCCTCAGAGGCCCATGTTAGAGGATGACACAGCTATTGACCTATTTCTACCTTTCTTCTTCATCTACAGGCTCAGAAGTATCATCTGTCTTCATCTTCCCCCCAAAGACCAAAGATGTGCTCACCATCACTCTGACTCCTAAGGTCACGTGTGTTGTGGTAGACATTAGCCAGAATGATCCCGAGGTCCGGTTCAGCTGGTTTATAGATGACGTGGAAGTCCACACAGCTCAGACTCATGCCCCGGAGAAGCAGTCCAACAGCACTTTACGCTCAGTCAGTGAACTCCCCATCGTGCACCGGGACTGGCTCAATGGCAAGACGTTCAAATGCAAAGTCAACAGTGGAGCATTCCCTGCCCCCATCGAGAAAAGCATCTCCAAACCCGAAGGTGGGAGCAGCAGGGTGTGTGGTGTAGAAGCTGCAGTAGGCCATAGACAGAGCTTGACTTAACTAGACTT |
| 5′ HA m1_HC (mIgG1 WT > Fab’ fragment) | CAGTATTGTCCAGATTGTGTGCAGCCATATGGCCCAGGTATAAGAGGTTTAACAGTGGAACACAGATGCCCACATCAGACAGCTGGGGGGCGGGGGTGAACACAGATACCCATACTGGAAAGCAGGTGGGGCATTTTCCTAGGAACGGGACTGGGCTCAATGGCCTCAGGTCTCATCTGGTCTGGTGATCCTGACATTGATAGGCCCAAATGTTGGATATCACCTACTCCATGTAGAGAGTCGGGGACATGGGAAGGGTGCAAAAGAGCGGCCTTCTAGAAGGTTTGGTCCTGTCCTGTCCTGTCTGACAGTGTAATCACATATACTTTTTCTTGTAGCCAAAACGACACCCCCATCTGTCTATCCACTGGCCCCTGGATCTGCTGCCCAAACTAACTCCATGGTGACCCTGGGATGCCTGGTCAAGGGCTATTTCCCTGAGCCAGTGACAGTGACCTGGAACTCTGGATCCCTGTCCAGCGGTGTGCACACCTTCCCAGCTGTCCTGCAGTCTGACCTCTACACTCTGAGCAGCTCAGTGACTGTCCCCTCCAGCACCTGGCCCAGCCAGACCGTCACCTGCAACGTTGCTCATCCGGCATGTAGCACCAAGGTAGACAAAAAGATA |
| 3′ HA m1_HC (mIgG1 WT> Fab’ fragment) | GGTGAGAGGACGTATAGGGAGGAGGGGTTCACTAGAGGTGAGGCTCAAGCCATTAGCCTGCCTAAACCAACCAGGCTGGACAGCCATCACCAGGAAATGGATCTCAGCCCAGAAGATCGAAAGTTGTTCTTCTCCCTTCTGGAGATTTCTATGTCCTTTACACTCATTGGTTAATATCCTGGGTTGGATTCCCACACATCTTGACAAACAGAGACAATTGAGTATCACCAGCCAAAAGTCATACCCAAAAACAGCCTGGCATGACCTCACACCAGACTCAAACTTACCCTACCTTTATCCTGGTGGCTTCTCATCTCCAGACCCCAGTAACACATAGCTTTCTCTCCACAGTGCCCAGGGATTGTGGTTGTAAGCCTTGCATATGTACAGGTAAGTCAGTAGGCCTTTCACCCTGACCCCAGATGCAACAAGTGGCCATGTTAGAGGGTGGCCCAGGTATTGACCTATTTCCACCTTTCTTCTTCATCCTTAGTCCCAGAAGTATCATCTGTCTTCATCTTCCCCCCAAAGCCCAAGGATGTGCTCACCATTACTCTGACTCCTAAGGTCACGTGTGTTGTGGTAGACATCAGCAAGGATGATCCCGAGGTCCAGTTCAGCTGGTTTGT |
| 5′ HA mκ_LC (mκ LC > mκLC-tag) | TCTGTCTGAAGCATGGAACTGAAAAGAATGTAGTTTCAGGGAAGAAAGGCAATAGAAGGAAGCCTGAGAATATCTTCAAAGGGTCAGACTCAATTTACTTTCTAAAGAAGTAGCTAGGAACTAGGGAATAACTTAGAAACAACAAGATTGTATATATGTGCATCCTGGCCCCATTGTTCCTTATCTGTAGGGATAAGCGTGCTTTTTTGTGTGTCTGTATATAACATAACTGTTTACACATAATACACTGAAATGGAGCCCTTCCTTGTTACTTCATACCATCCTCTGTGCTTCCTTCCTCAGGGGCTGATGCTGCACCAACTGTATCCATCTTCCCACCATCCAGTGAGCAGTTAACATCTGGAGGTGCCTCAGTCGTGTGCTTCTTGAACAACTTCTACCCCAAAGACATCAATGTCAAGTGGAAGATTGATGGCAGTGAACGACAAAATGGCGTCCTGAACAGTTGGACTGATCAGGACAGCAAAGACAGCACCTACAGCATGAGCAGCACCCTCACGTTGACCAAGGACGAGTATGAACGACATAACAGCTATACCTGTGAGGCCACTCACAAGACATCAACTTCACCGATTGTAAAGAGTTTTAATAGCAATGAATGCGGATCC |
| 3′ HA mκ_LC (mκ LC > mκLC-tag) | TGAAGACAAAGATCCTGAGACGCCACCACCAGCTCCCCAGCTCCATCCTATCTTCCCTTCTAAGGTCTTGGAGGCTTCCCCACAAGCGACCTACCACTGTTGCGGTGCTCCAAACCTCCTCCCCACCTCCTTCTCCTCCTCCTCCCTTTCCTTGGCTTTTATCATGCTAATATTTGCAGAAAATATTCAATAAAGTGAGTCTTTGCACTTGAGATCTCTGTCTTTCTTACTAAATGGTAGTAATCAGTTGTTTTTCCAGTTACCTGGGTTTCTCTTCTAAAGAAGTTAAATGTTTAGTTGCCCTGAAATCCACCACACTTAAAGGATAAATAAAACCCTCCACTTGCCCTGGTTGGCTGTCCACTACATGGCAGTCCTTTCTAAGGTTCACGAGTACTATTCATGGCTTATTTCTCTGGGCCATGGTAGGTTTGAGGAGGCATACTTCCTAGTTTTCTTCCCCTAAGTCGTCAAAGTCCTGAAGGGGGACAGTCTTTACAAGCACATGTTCTGTAATCTGATTCAACCTACCCAGTAAACTTGGCGAAGCAAAGTAGAATCATTATCACAGGAAGCAAAGGCAACCTAAATGTGCA |
配方
- 洗涤缓冲液
PBS + 0.5% BSA - PBA
PBS + 5% BSA + 0.03% NaN3 - 培养基
RPMI-1640(含HEPES)+ 10%热灭活FBS + 2 mM超极性谷氨酰胺-1 + 1%(v/v)抗生素-抗真菌 + 新鲜添加的50 μM β-ME。 - 1×选择培养基
RPMI-1640(含HEPES)+ 10%热灭活FBS + 2 mM超极性谷氨酰胺 + 1%(v/v)抗生素-抗真菌 + 新鲜添加的50 μM β-ME + 新鲜添加的选择性抗生素,浓度预先确定。
References
- Adhikari, P., Zacharias, N., Ohri, R. and Sadowsky, J. (2020). Site-Specific Conjugation to Cys-Engineered THIOMABTM Antibodies. In: Tumey, L. (Ed.). Antibody-Drug Conjugates. Methods in Molecular Biology. pp. 51-69.
- Beers, S. A., Glennie, M. J. and White, A. L. (2016). Influence of immunoglobulin isotype on therapeutic antibody function. Blood 127(9): 1097-1101.
- Cong, L., Ran, F. A., Cox, D., Lin, S., Barretto, R., Habib, N., Hsu, P. D., Wu, X., Jiang, W., Marraffini, L. A., et al. (2013). Multiplex Genome Engineering Using CRISPR/Cas Systems. Science 339(6121): 819-823.
- Evers, M., Stip, M., Keller, K., Willemen, H., Nederend, M., Jansen, M., Chan, C., Budding, K., Nierkens, S., Valerius, T., et al. (2021). Anti-GD2 IgA kills tumors by neutrophils without antibody-associated pain in the preclinical treatment of high-risk neuroblastoma. J Immunother Cancer 9(10): e003163.
- Fennemann, F. L., Le Gall, C. M., van der Schoot, J. M. S., Ramos-Tomillero, I., Figdor, C. G., Scheeren, F. A., and Verdoes, M. (2021). Generation of dendritic cell targeted anti-cancer vaccines by CRISPR- Cas9-editing of the hybridoma immunoglobulin locus. In: Molecularly Defined Dendritic Cell-Targeted Vaccines for Cancer Immunotherapy. pp. 42-63.
- Le Gall, C. M., van der Schoot, J. M. S., Ramos-Tomillero, I., Khalily, M. P., van Dalen, F. J., Wijfjes, Z., Smeding, L., van Dalen, D., Cammarata, A., Bonger, K. M., et al. (2021). Dual Site-Specific Chemoenzymatic Antibody Fragment Conjugation Using CRISPR-Based Hybridoma Engineering. Bioconjug Chem 32(2): 301-310.
- Khoshnejad, M., Brenner, J. S., Motley, W., Parhiz, H., Greineder, C. F., Villa, C. H., Marcos-Contreras, O. A., Tsourkas, A. and Muzykantov, V. R. (2018). Molecular engineering of antibodies for site-specific covalent conjugation using CRISPR/Cas9. Sci Rep 8(1): 1760.
- Kim, H. K., Lee, S., Kim, Y., Park, J., Min, S., Choi, J. W., Huang, T. P., Yoon, S., Liu, D. R. and Kim, H. H. (2020). High-throughput analysis of the activities of xCas9, SpCas9-NG and SpCas9 at matched and mismatched target sequences in human cells. Nat Biomed Eng 4(1): 111-124.
- Kirkling, M. E., Cytlak, U., Lau, C. M., Lewis, K. L., Resteu, A., Khodadadi-Jamayran, A., Siebel, C. W., Salmon, H., Merad, M., Tsirigos, A., et al. (2018). Notch Signaling Facilitates In Vitro Generation of Cross-Presenting Classical Dendritic Cells. Cell Rep 23(12): 3658-3672 e3656.
- Köhler, G. and Milstein, C. (1975) Continuous cultures of fused cells secreting antibody of predefined specificity. Nature 256(5517): 495-497.
- Nanda, J.S. and Lorsch, J.R. (2014). Labeling a protein with fluorophores using NHS ester derivitization. Methods Enzymol 536: 87-94.
- van der Schoot, J. M. S., Fennemann, F. L., Valente, M., Dolen, Y., Hagemans, I. M., Becker, A. M. D., Le Gall, C. M., van Dalen, D., Cevirgel, A., van Bruggen, J. A. C., et al. (2019). Functional diversification of hybridoma-produced antibodies by CRISPR/HDR genomic engineering. Sci Adv 5(8): eaaw1822.
- Sharma, A., Subudhi, S. K., Blando, J., Scutti, J., Vence, L., Wargo, J., Allison, J. P., Ribas, A., and Sharma, P. (2019). Anti-CTLA-4 immunotherapy does not deplete FOXP3+ regulatory T cells (Tregs) in human cancers.Clin Cancer Res 25(4): 1233-1238.
- Waldor, M. K., Mitchell, D., Kipps, T. J., Herzenberg, L. A., and Steinman, L. (1987). Importance of immunoglobulin isotype in therapy of experimental autoimmune encephalomyelitis with monoclonal anti-CD4 antibody. J Immunol 139(11): 360-3664.
- Yamada, K., Shikida, N., Shimbo, K., Ito, Y., Khedri, Z., Matsuda, Y. and Mendelsohn, B. A. (2019). AJICAP: Affinity Peptide Mediated Regiodivergent Functionalization of Native Antibodies. Angew Chem Int Ed Engl 58(17): 5592-5597.
- Zhao, Y., Gutshall, L., Jiang, H., Baker, A., Beil, E., Obmolova, G., Carton, J., Taudte, S. and Amegadzie, B. (2009). Two routes for production and purification of Fab fragments in biopharmaceutical discovery research: Papain digestion of mAb and transient expression in mammalian cells. Protein Expr Purif 67(2): 182-189.
- 本文作者: Anderson
- 本文链接: http://nikolahuang.github.io/2024/03/14/的免疫球蛋白位点工程/
- 版权声明: 转载请注明出处,谢谢。

